2. Les tableaux numpy.array#
Le module numpy permet de manipuler les tableaux de données, données qui ont toutes le même type numérique. Le nombre de dimension est variable. Ce module est particulièrement optimisé et est au centre de tous les modules de calculs scientifiques en python. Il faut donc connaitre ce module et savoir utiliser la structure numpy.array.
La syntaxe introduite dans numpy est devenue une référence et est utilisée dans les autres modules tels que pytorch ou tensorflow. Ces modules spécialisés dans le deep learning ont recodé les tableaux (appelés tensor) pour pouvoir être utilisés sur GPU (carte graphique) et faire du calcul intensif. Ce n’est pas le cas des numpy.array.
import numpy as np
2.1. Création et propriétés#
Création à partir d’une liste de listes. Sans option supplémentaire le type est détecté automatiquement.
a = np.array([[8,3,2,4], [5,1,6,0], [9,7,4,1]])
print(a)
[[8 3 2 4]
[5 1 6 0]
[9 7 4 1]]
print("L'objet a est de type:", type(a))
print(f"L'appel a[0] vaut {a[0]} et est de type", type(a[0]))
print(f"L'appel a[0,0] vaut {a[0,0]} et est de type", type(a[0,0]))
L'objet a est de type: <class 'numpy.ndarray'>
L'appel a[0] vaut [8 3 2 4] et est de type <class 'numpy.ndarray'>
L'appel a[0,0] vaut 8 et est de type <class 'numpy.int64'>
Remarque: le type entier utilisé dans numpy est le type numpy.int64 c’est à dire un entier codé sur 64 bits et le nombre le type int par défaut de python.
Quelques champs qui informent sur la structure du tableau a.
print("Dimension:", a.ndim)
print("Taille:", a.size) # attention len(a) = 3
print("Forme:", a.shape)
print("Types des éléments:", a.dtype) # type des éléments
Dimension: 2
Taille: 12
Forme: (3, 4)
Types des éléments: int64
a.sum() == np.sum(a) # on peut utiliser la méthode ou une fonction globale
True
Pour changer le type des éléments on peut utiliser la méthode astype:
a.astype('complex')
array([[8.+0.j, 3.+0.j, 2.+0.j, 4.+0.j],
[5.+0.j, 1.+0.j, 6.+0.j, 0.+0.j],
[9.+0.j, 7.+0.j, 4.+0.j, 1.+0.j]])
a.astype('float').tolist() # bien sûr on peut enchaîner les méthodes
[[8.0, 3.0, 2.0, 4.0], [5.0, 1.0, 6.0, 0.0], [9.0, 7.0, 4.0, 1.0]]
2.2. Création à partir de fonctions numpy#
np.array([ i+1 for i in range(10) ])
array([ 1, 2, 3, 4, 5, 6, 7, 8, 9, 10])
np.array([ i+1 for i in range(10) ], dtype='float', ndmin=2)
array([[ 1., 2., 3., 4., 5., 6., 7., 8., 9., 10.]])
np.arange(10, 20, step=2) # même syntaxe que range
array([10, 12, 14, 16, 18])
Remarque: pour la fonction np.arange on a effectivement création en mémoire de la plage de valeurs alors que range renvoie un objet abstrait itérable (pas de création mémoire).
np.zeros((2, 4)) # de même il y a np.ones
array([[0., 0., 0., 0.],
[0., 0., 0., 0.]])
La matrice indicatrice s’obtient par la méthode eye.
np.eye(3)
array([[1., 0., 0.],
[0., 1., 0.],
[0., 0., 1.]])
np.diag([1., 2, 3])
array([[1., 0., 0.],
[0., 2., 0.],
[0., 0., 3.]])
np.linspace(0, 1, num=6)
array([0. , 0.2, 0.4, 0.6, 0.8, 1. ])
Il faut toujours consuler l’aide disponible. Par exemple consulter la documentation de la fonction np.asarray.
2.3. Dimensions et axes#
En numpy les tableaux multidimensionnels sont contruits en utilisant la représentation en ligne (row-major order, cf. page wikipedia).
Ainsi un tableau de dimension 1 peut s’identifier comme un vecteur ligne.
Une matrice (dimension 2) peut se voir comme un tableau de lignes.
u = np.array([1., 4, 10])
a = np.array([[8., 3, 2], [5, 1., 6]])
print("une vecteur ligne: ", u)
print("une matrice: ", a)
une vecteur ligne: [ 1. 4. 10.]
une matrice: [[8. 3. 2.]
[5. 1. 6.]]
u.dtype
dtype('float64')
Le vecteur ligne u a 1 seule dimension, 1 seul axe (axis) de taille 3.
La matrice a a 2 dimensions, 2 axes. Le premier axe (axis 0) est de taille 2 et le deuxième axe est de taille 3.
a[0, 1] # on accède à l'élément d'indice 0 sur le 1er axe et et d'indice 1 sur le 2ème axe
3.0
2.4. Accès aux éléments#


2.5. Modification, transformation#
La méthode
reshaperenvoie une vue du tableau qui l’appelle et ce tableau n’est pas modifié.La méthode
resizemodifie le tableau (change la forme)
a = np.arange(8)
b = a.reshape((2,4)) # renvoie **une vue de a**, a et b partagent les mêmes données
print("a:", a)
print("b:", b)
a: [0 1 2 3 4 5 6 7]
b: [[0 1 2 3]
[4 5 6 7]]
b[1,1] = 100 # on modifie b: l'élément 5 devient 100
print("a:", a) # on affiche a
a: [ 0 1 2 3 4 100 6 7]
b.resize((4,2)) # modification in-place
print("b:", b)
print("a:", a) # a n'est pas modifié
b: [[ 0 1]
[ 2 3]
[ 4 100]
[ 6 7]]
a: [ 0 1 2 3 4 100 6 7]
Pour ajouter un axe, par exemple pour transformer un vecteur ligne en vecteur colonne on peut utiliser la méthode reshape ou ajouter un axe avec la syntaxe [:, np.newaxis] ou [:, None].
a.T
array([ 0, 1, 2, 3, 4, 100, 6, 7])
a.reshape(8, 1)
a[:, np.newaxis] # un vecteur ligne transformé en vecteur colonne
array([[ 0],
[ 1],
[ 2],
[ 3],
[ 4],
[100],
[ 6],
[ 7]])
a[:, None] # le mot clé np.newaxis est équivalent à None
array([[ 0],
[ 1],
[ 2],
[ 3],
[ 4],
[100],
[ 6],
[ 7]])
for i,x in enumerate(b.flat): # b.flat version "vue" applatie de b
print(i, x)
0 0
1 1
2 2
3 3
4 4
5 100
6 6
7 7
c = b.flatten() # une copie applatie (tous les axes sont écrasés)
c[1] = 50
c
array([ 0, 50, 2, 3, 4, 100, 6, 7])
b
array([[ 0, 1],
[ 2, 3],
[ 4, 100],
[ 6, 7]])
a
array([ 0, 1, 2, 3, 4, 100, 6, 7])
2.6. Opérations#
print("moyenne: ", np.mean(a)) # moyenne
print("variance: ", np.var(a)) # version biaisée voir documentation
print("variance sans biais: ", np.var(a, ddof=1)) # version sans biais
moyenne: 15.375
variance: 1027.984375
variance sans biais: 1174.8392857142858
import math
# math.sqrt(a) # erreur si on execute cette cellule
np.sqrt(a) # toujours utiliser les fonctions numpy
array([ 0. , 1. , 1.41421356, 1.73205081, 2. ,
10. , 2.44948974, 2.64575131])
np.maximum(a, 4)
array([ 4, 4, 4, 4, 4, 100, 6, 7])
np.max(a)
100
2.7. Opérations par axe, option axis#
Les opérations par axe sont très importantes et doivent être bien comprises.
print(f"{b.shape[0]} lignes (axe 0) et {b.shape[1]} colonnes (axe 1)")
print(b)
b
4 lignes (axe 0) et 2 colonnes (axe 1)
[[ 0 1]
[ 2 3]
[ 4 100]
[ 6 7]]
array([[ 0, 1],
[ 2, 3],
[ 4, 100],
[ 6, 7]])
np.sum(b)
123
np.sum(b, axis = 0) # agit sur l'axe 0: on réduit l'axe 0 -> la somme des lignes
array([ 12, 111])
np.sum(b, axis = 1) # agit sur l'axe 1: on réduit l'axe 1 -> la somme des colonnes
array([ 1, 5, 104, 13])
np.max(b, axis = 0)
array([ 6, 100])
2.7.1. Exercice#
On veut calculer
pour différentes valeurs de \(n\), \(n=10,100,1000,10000,100000\).
Ecrire 2 implémentations différentes: l’une un python sans
numpyet l’autre en utilisantnumpy.Comparer les temps de calculs de ces 2 versions.
def sum_squares_python(n):
result = 0
for k in range(n+1):
result += k**2
return result
def sum_squares_numpy(n):
z = np.arange(n+1)
result = np.sum(z**2)
return result
for n in 10**np.arange(5):
print(n)
%timeit sum_squares_python(n)
Show code cell output
1
149 ns ± 2.21 ns per loop (mean ± std. dev. of 7 runs, 10,000,000 loops each)
10
361 ns ± 3.03 ns per loop (mean ± std. dev. of 7 runs, 1,000,000 loops each)
100
2.96 μs ± 4.91 ns per loop (mean ± std. dev. of 7 runs, 100,000 loops each)
1000
32 μs ± 361 ns per loop (mean ± std. dev. of 7 runs, 10,000 loops each)
10000
362 μs ± 1.52 μs per loop (mean ± std. dev. of 7 runs, 1,000 loops each)
for n in 10**np.arange(5):
print(n)
%timeit sum_squares_numpy(n)
Show code cell output
1
1.66 μs ± 20.2 ns per loop (mean ± std. dev. of 7 runs, 1,000,000 loops each)
10
1.87 μs ± 252 ns per loop (mean ± std. dev. of 7 runs, 100,000 loops each)
100
1.76 μs ± 7.78 ns per loop (mean ± std. dev. of 7 runs, 1,000,000 loops each)
1000
---------------------------------------------------------------------------
KeyboardInterrupt Traceback (most recent call last)
Cell In[42], line 3
1 for n in 10**np.arange(5):
2 print(n)
----> 3 get_ipython().run_line_magic('timeit', 'sum_squares_numpy(n)')
File ~/.venv/lib/python3.11/site-packages/IPython/core/interactiveshell.py:2480, in InteractiveShell.run_line_magic(self, magic_name, line, _stack_depth)
2478 kwargs['local_ns'] = self.get_local_scope(stack_depth)
2479 with self.builtin_trap:
-> 2480 result = fn(*args, **kwargs)
2482 # The code below prevents the output from being displayed
2483 # when using magics with decorator @output_can_be_silenced
2484 # when the last Python token in the expression is a ';'.
2485 if getattr(fn, magic.MAGIC_OUTPUT_CAN_BE_SILENCED, False):
File ~/.venv/lib/python3.11/site-packages/IPython/core/magics/execution.py:1199, in ExecutionMagics.timeit(self, line, cell, local_ns)
1196 if time_number >= 0.2:
1197 break
-> 1199 all_runs = timer.repeat(repeat, number)
1200 best = min(all_runs) / number
1201 worst = max(all_runs) / number
File /opt/homebrew/Cellar/python@3.11/3.11.11/Frameworks/Python.framework/Versions/3.11/lib/python3.11/timeit.py:208, in Timer.repeat(self, repeat, number)
206 r = []
207 for i in range(repeat):
--> 208 t = self.timeit(number)
209 r.append(t)
210 return r
File ~/.venv/lib/python3.11/site-packages/IPython/core/magics/execution.py:173, in Timer.timeit(self, number)
171 gc.disable()
172 try:
--> 173 timing = self.inner(it, self.timer)
174 finally:
175 if gcold:
File <magic-timeit>:1, in inner(_it, _timer)
Cell In[40], line 3, in sum_squares_numpy(n)
1 def sum_squares_numpy(n):
2 z = np.arange(n+1)
----> 3 result = np.sum(z**2)
4 return result
File ~/.venv/lib/python3.11/site-packages/numpy/core/fromnumeric.py:2313, in sum(a, axis, dtype, out, keepdims, initial, where)
2310 return out
2311 return res
-> 2313 return _wrapreduction(a, np.add, 'sum', axis, dtype, out, keepdims=keepdims,
2314 initial=initial, where=where)
File ~/.venv/lib/python3.11/site-packages/numpy/core/fromnumeric.py:72, in _wrapreduction(obj, ufunc, method, axis, dtype, out, **kwargs)
71 def _wrapreduction(obj, ufunc, method, axis, dtype, out, **kwargs):
---> 72 passkwargs = {k: v for k, v in kwargs.items()
73 if v is not np._NoValue}
75 if type(obj) is not mu.ndarray:
76 try:
File ~/.venv/lib/python3.11/site-packages/numpy/core/fromnumeric.py:72, in <dictcomp>(.0)
71 def _wrapreduction(obj, ufunc, method, axis, dtype, out, **kwargs):
---> 72 passkwargs = {k: v for k, v in kwargs.items()
73 if v is not np._NoValue}
75 if type(obj) is not mu.ndarray:
76 try:
KeyboardInterrupt:
2.8. Broadcasting#
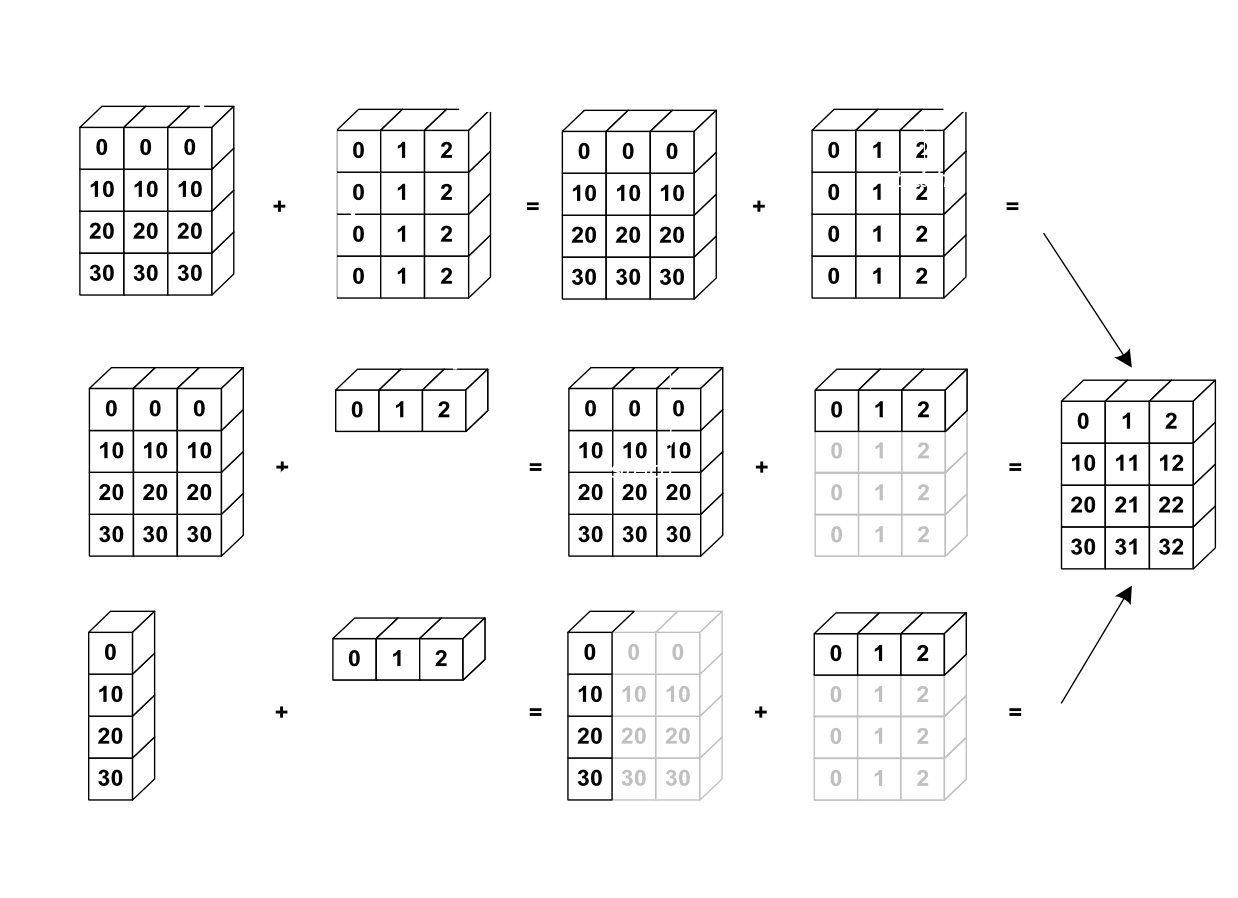
Mécanisme très utilisé qu’il faut connaitre pour une bonne utilisation! Attention il peut y avoir des pièges.
a = 10 * np.arange(4) # ou bien np.arange(31, step=10)
b = np.arange(3)
print("a =", a)
print("b =", b)
a = [ 0 10 20 30]
b = [0 1 2]
c = a[:, np.newaxis]
print("c =", c)
c = [[ 0]
[10]
[20]
[30]]
print(c + b) # broadcasting
[[ 0 1 2]
[10 11 12]
[20 21 22]
[30 31 32]]
Pour une utilisation avancée du broadcasting (par exemple pour appliquer une fonction qui n’est pas un opérateur arithmétique classique) on peut utiliser np.broadcast
x = np.array([[1], [2], [3]])
y = np.array([40, 50, 60])
b = np.broadcast(x, y)
for u, v in b:
print(u, v)
1 40
1 50
1 60
2 40
2 50
2 60
3 40
3 50
3 60
b = np.broadcast(x, y)
out = np.empty(b.shape)
out.flat = [u+v for (u,v) in b]
out
array([[41., 51., 61.],
[42., 52., 62.],
[43., 53., 63.]])
2.8.1. Exercice#
Construire la matrice
de plusieurs façons différentes:
1. construire un vecteur ligne (1 seul axe) \(u= \begin{pmatrix} 0 & 10 & 20 & 30 \end{pmatrix}\) puis le convertir en vecteur colonne \(v = u^\top\) en utilisant la méthode reshape ou l’ajout d’un axe via np.newaxis, et appeler la fonction np.repeat
2. construire directement le vecteur colonne \(v\) via l’option ndim=2 et la fonction np.repeat
3. construire le vecteur ligne \(u\) puis utiliser np.repeat et ensuite la méthode reshape.
# construction 1.
u = np.arange(4)*10 # vecteur ligne (1 seul axe 0)
# deux options pour transformer ce vecteur ligne en vecteur colonne:
u.reshape((4, 1)) # on change la forme avec la méthode reshape
u[:, None] # on ajoute un axe supplémentaire avec cette syntaxe
# pour plus de lisibilité on peut remplacer le mot-clé None par np.newaxis (c'est pareil!)
v = u[:, np.newaxis] # écriture un peu plus explicite
# une fois qu'on a un vecteur colonne, on fait une répétition
np.repeat(v, 3, axis=1) # on agit sur l'axe 1 pour faire la répétition
# construction 2.
# il est possible d'utiliser l'option ndmin=2
v = np.array(np.arange(0, 40, step=10), ndmin=2)
v = np.array([i for i in range(0,40,10)], ndmin=2)
np.repeat(v, 3, axis=1) # on agit sur l'axe 1 pour faire la répétition
# construction 3.
u = np.arange(0, 40, step=10)
np.repeat(u, 3).reshape((4, 3))
Show code cell output
array([[ 0, 0, 0],
[10, 10, 10],
[20, 20, 20],
[30, 30, 30]])
2.9. Algèbre linéaire#
En bref:
innerpour calculer le produit scalaire, on peut aussi utiliserdotouvdotouterpour produit tensoriel entre 2 vecteursopérateur
@pour un produit matriciel, ou la fonctionmatmulpour la transposée d’une matrice
aon utilisea.Teinsumfonction très générale et très optimisée, lire la documention…
Les transformations sont dans le sous-module linalg de numpy
choleskyfactorisation de Choleskyqrdécomposition QRsvddécomposition en valeurs singulièresinvinverse une matricesolverésolution d’un système linéaire
Pour en savoir plus il y a:
print(dir(np.linalg))
['LinAlgError', '__all__', '__builtins__', '__cached__', '__doc__', '__file__', '__loader__', '__name__', '__package__', '__path__', '__spec__', '_umath_linalg', 'cholesky', 'cond', 'det', 'eig', 'eigh', 'eigvals', 'eigvalsh', 'inv', 'linalg', 'lstsq', 'matrix_power', 'matrix_rank', 'multi_dot', 'norm', 'pinv', 'qr', 'slogdet', 'solve', 'svd', 'tensorinv', 'tensorsolve', 'test']
u = np.arange(4)
v = 2+u
print("u =", u, "\nv =", v)
u = [0 1 2 3]
v = [2 3 4 5]
np.inner(u, v) == np.sum(u*v)
True
A = np.outer(u, v)
print(A)
[[ 0 0 0 0]
[ 2 3 4 5]
[ 4 6 8 10]
[ 6 9 12 15]]
print("attention au produit: \n", A * u)
print("voici le produit matrice-vecteur: \n", A @ u)
attention au produit:
[[ 0 0 0 0]
[ 0 3 8 15]
[ 0 6 16 30]
[ 0 9 24 45]]
voici le produit matrice-vecteur:
[ 0 26 52 78]
A.T @ u
array([28, 42, 56, 70])
