Liste des modules python utilisés
import numpy as np
import scipy.stats as sps
import matplotlib.pyplot as plt
import seaborn as sns
sns.set_theme()import numpy as np
import scipy.stats as sps
import matplotlib.pyplot as plt
import seaborn as sns
sns.set_theme()random de NumPyLe sous-module random de NumPy est utilisé pour générer des nombres pseudo-aléatoires et des échantillons à partir de distributions statistiques communes (normal, uniforme, poisson, etc.).
Le sous-module random de NumPy a subi une refonte significative dans ses versions récentes. La version 1.17 (2019) introduit un nouveau mécanisme pour la génération de nombres pseudo-aléatoires. Ce mécanisme repose sur un objet appelé Generator, remplaçant les fonctions globales antérieures de NumPy (le contexte globale était appelé RandomState). Plutôt que d’appeler directement des fonctions globales comme np.random.normal ou np.random.uniform, il est maintenant préférable d’instancier un objet Generator, et d’appeler ses méthodes pour générer des réalisations de variables aléatoires.
Il est recommandé de lire la documentation officielle. Par défaut l’algorithme utilisé pour les nombres pseudo-aléatoires est le PCG64.
Voici un exemple d’utilisation de ce mécanisme basé sur l’objet Generator.
from numpy.random import default_rng, SeedSequence
sq = SeedSequence()
seed = sq.entropy # on sauve la graine pour reproduire les résultats
rng = default_rng(sq)
rng.standard_normal(5)array([-1.48056146, -1.16460962, -0.59118793, -0.66373832, 0.50993954])Dans le code précédent:
SeedSequence crée une séquence de graine qui produit une graine de manière déterministe ou aléatoire. Sans paramètre, SeedSequence() utilise l’état aléatoire global de la machine pour générer une graine.rng est un objet Generator qui produit des nombres pseudo-aléatoires selon l’algorithme PCG64,Pour reproduire les résultats, on initialise un nouvel objet rng2 avec la graine sauvegardée.
rng2 = default_rng(seed)
rng2.standard_normal(10)array([-1.48056146, -1.16460962, -0.59118793, -0.66373832, 0.50993954,
-0.73029163, 0.02509922, 0.89766102, 0.42894541, -0.91778999])L’objet SeedSequence permet aussi d’initialiser plusieurs Generator considérés comme indépendants: c’est important pour écrire un code parallèle (multithreading).
stats de SciPyLe module scipy.stats utilise une programmation type orientée objet.
La principale utilisation de ce module est la manipulation de variables aléatoires. On initialise un objet via le nom d’une distribution. Puis de cet objet on peut connaitre la densité/masse, la fonction de répartition ou obtenir des réalisations (en fait obtenues généralement via NumPy).
dist_continu = [d for d in dir(sps) if
isinstance(getattr(sps, d), sps.rv_continuous)]
dist_discrete = [d for d in dir(sps) if
isinstance(getattr(sps, d), sps.rv_discrete)]
print("Nombres de distributions à densité: ", len(dist_continu))
print(dist_continu)
print("Nombres de distributions discrètes: ", len(dist_discrete))
print(dist_discrete)Nombres de distributions à densité: 110
['alpha', 'anglit', 'arcsine', 'argus', 'beta', 'betaprime', 'bradford', 'burr', 'burr12', 'cauchy', 'chi', 'chi2', 'cosine', 'crystalball', 'dgamma', 'dpareto_lognorm', 'dweibull', 'erlang', 'expon', 'exponnorm', 'exponpow', 'exponweib', 'f', 'fatiguelife', 'fisk', 'foldcauchy', 'foldnorm', 'gamma', 'gausshyper', 'genexpon', 'genextreme', 'gengamma', 'genhalflogistic', 'genhyperbolic', 'geninvgauss', 'genlogistic', 'gennorm', 'genpareto', 'gibrat', 'gompertz', 'gumbel_l', 'gumbel_r', 'halfcauchy', 'halfgennorm', 'halflogistic', 'halfnorm', 'hypsecant', 'invgamma', 'invgauss', 'invweibull', 'irwinhall', 'jf_skew_t', 'johnsonsb', 'johnsonsu', 'kappa3', 'kappa4', 'ksone', 'kstwo', 'kstwobign', 'landau', 'laplace', 'laplace_asymmetric', 'levy', 'levy_l', 'levy_stable', 'loggamma', 'logistic', 'loglaplace', 'lognorm', 'loguniform', 'lomax', 'maxwell', 'mielke', 'moyal', 'nakagami', 'ncf', 'nct', 'ncx2', 'norm', 'norminvgauss', 'pareto', 'pearson3', 'powerlaw', 'powerlognorm', 'powernorm', 'rayleigh', 'rdist', 'recipinvgauss', 'reciprocal', 'rel_breitwigner', 'rice', 'semicircular', 'skewcauchy', 'skewnorm', 'studentized_range', 't', 'trapezoid', 'triang', 'truncexpon', 'truncnorm', 'truncpareto', 'truncweibull_min', 'tukeylambda', 'uniform', 'vonmises', 'vonmises_line', 'wald', 'weibull_max', 'weibull_min', 'wrapcauchy']
Nombres de distributions discrètes: 21
['bernoulli', 'betabinom', 'betanbinom', 'binom', 'boltzmann', 'dlaplace', 'geom', 'hypergeom', 'logser', 'nbinom', 'nchypergeom_fisher', 'nchypergeom_wallenius', 'nhypergeom', 'planck', 'poisson', 'poisson_binom', 'randint', 'skellam', 'yulesimon', 'zipf', 'zipfian']Voici un exemple d’utilisation aver la loi exponentielle (de paramètre \(\lambda = 2\)).
lambd = 2
E = sps.expon(scale = 1./lambd)
E.cdf(1) # appel de la méthode `cdf` fonction de répartition
fig, (ax1, ax2) = plt.subplots(ncols=2, nrows=1)
x = np.linspace(E.ppf(0.01), E.ppf(0.99), 100)
ax1.plot(x, E.pdf(x))
ax1.set_title("Densité")
ax2.plot(x, E.cdf(x), color='C1')
ax2.set_title("Fonction de répartition")
fig.suptitle(fr"Loi exponentielle de paramètre $\lambda={lambd}$");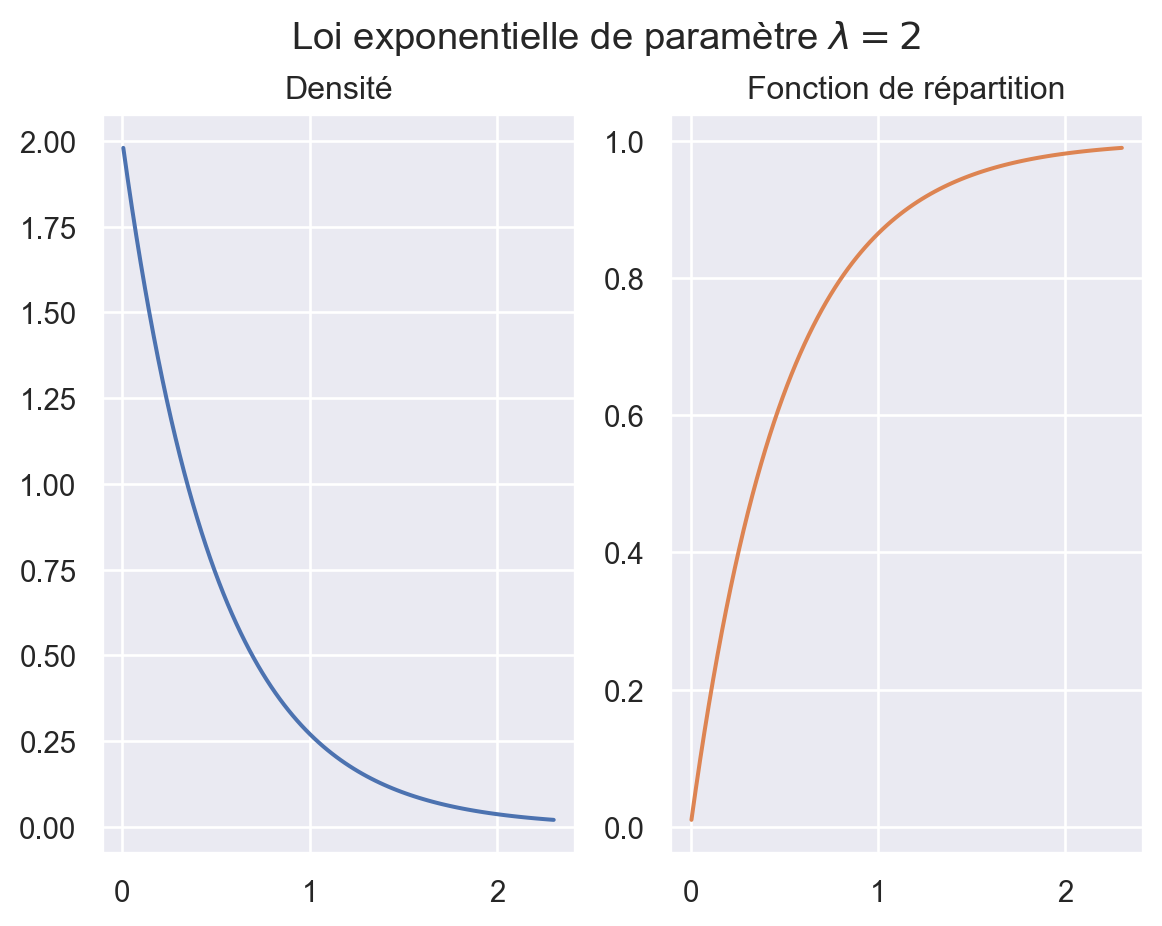
Pour obtenir des réalisations indépendantes de cette variable aléatoire \(E\) on appelle la méthode rvs. Par exemple
E.rvs(size=(3, 5), random_state=rng)array([[0.07169342, 0.59781183, 0.09937329, 0.07629887, 0.73913764],
[0.3211071 , 0.6045416 , 0.51018347, 1.14066807, 0.18600614],
[0.40450665, 0.10907444, 0.71233015, 0.17422795, 0.11647732]])Pour être cohérent avec la nouvelle approche NumPy il faut passer l’objet rng notre générateur de nombres pseudo-aléatoires comme argument à la méthode rvs.
Le mouvement Brownien est l’une des briques de base pour la construction de processus à temps continu. Une autre brique de base est le processus de Poisson pour prendre en compte d’éventuelle discontinuité. Pour ces processus il est possible de faire de la simulation exacte, c’est à dire sans erreur de discrétisation.
La simulation exacte d’un processus \((X_t)_{t \in [0,T]}\) consiste en la simulation du vecteur aléatoire \((X_{t_0}, \dots, X_{t_N})\) pour des instants donnés \(0 = t_0 \le \cdots \le t_N = T\). Souvent on découpe l’intervalle \([0, T]\) en \(N\) intervalles de même longueur c’est à dire en considérant \(N+1\) points \(t_k\) définis par \[t_k = k \frac{T}{N} \quad \forall k \in \{0,\dots,N\}.\]
On rappelle deux définitions possibles (et équivalentes) d’un mouvement Brownien standard \((B_t)_{t \ge 0}\).
Exercice: Ecrire l’algorithme de simulation de \((B_0, \dots, B_{t_N})\) en utilisant la définition 1. puis la défintion 2. et faire le lien.
D’après la définition 1. on simule d’abord les accroissements indépendants \[ B_{t_{k+1}} - B_{t_k} \sim \mathcal{N}(0, t_{k+1}-t_{k}) \] puis on reconstruit \(B_{t_k} = \displaystyle \sum_{j=0}^{k-1} (B_{t_{j+1}} - B_{t_j})\) (somme téléscopique).
A partir de \(N\) gaussiennes indépendantes \((G_k)_{k=1,\dots,N}\) on construit le vecteur \((B_{t_1},\dots,B_{t_N})\) par la récurrence: \[ B_{t_{k+1}} = B_{t_k} + \sqrt{t_{k+1} - t_k} G_{k+1}. \]
Pour simuler \(M\) trajectoires il faut \(M \times N\) réalisations indépendantes de gaussiennes. En NumPy, dans la mesure du possible, il faut tout simuler d’un bloc (un seul appel de rng.standard_normal) et mettre en mémoire les \(M \times N\) données.
brownian_1ddef brownian_1d(n_paths: int,
times: np.ndarray,
increments: bool = False,
rng: np.random.Generator = None) -> np.ndarray:
"""
Simule des trajectoires de mouvement brownien standard en 1D.
Args:
n_paths: Nombre de trajectoires à simuler.
times: tableau 1d (list, tuple ou np.ndarray) de taille `n_times`
qui contient les instants $t_k$.
increments: Si `True`, les accroissements sont retournés,
sinon ce sont les trajectoires.
rng: objet `Generator`, création si `None` (non recommandé).
Returns:
- si `increments=False`: np.ndarray de forme `(n_paths, n_times)`
contenant les trajectoires aux instants $t_k$.
- si `increments=True`: np.ndarray de forme `(n_paths, n_times-1)`
contenant les accroissements entre les instants $t_k$.
"""
# Initialisation du générateur pseudo-aléatoire si non fourni
if rng is None:
rng = np.random.default_rng()
# Calcul des accroissements (utilisation du broadcasting)
dt = np.diff(times)
dB = np.sqrt(dt) * rng.standard_normal((n_paths, len(dt)))
# Si les accroissements sont demandés
if increments:
return dB
# Construction des trajectoires browniennes (cumul des incréments)
paths = np.hstack([np.zeros((n_paths,1)), np.cumsum(dB, axis=1)])
return pathsVoici un code illustrant l’utilisation de cette fonction.
N, M = 100, 200
T = 1
times = np.arange(N+1)*(T / N)
paths = brownian_1d(M, times, rng=rng)
fig, ax = plt.subplots()
for path in paths:
ax.plot(times, path, color='C0', alpha=0.3)
ax.plot(times, paths[0,:], color='C1', lw=2, label='One path')
ax.legend()
ax.set_title(f"{M} standard Brownian motion paths with N={N}");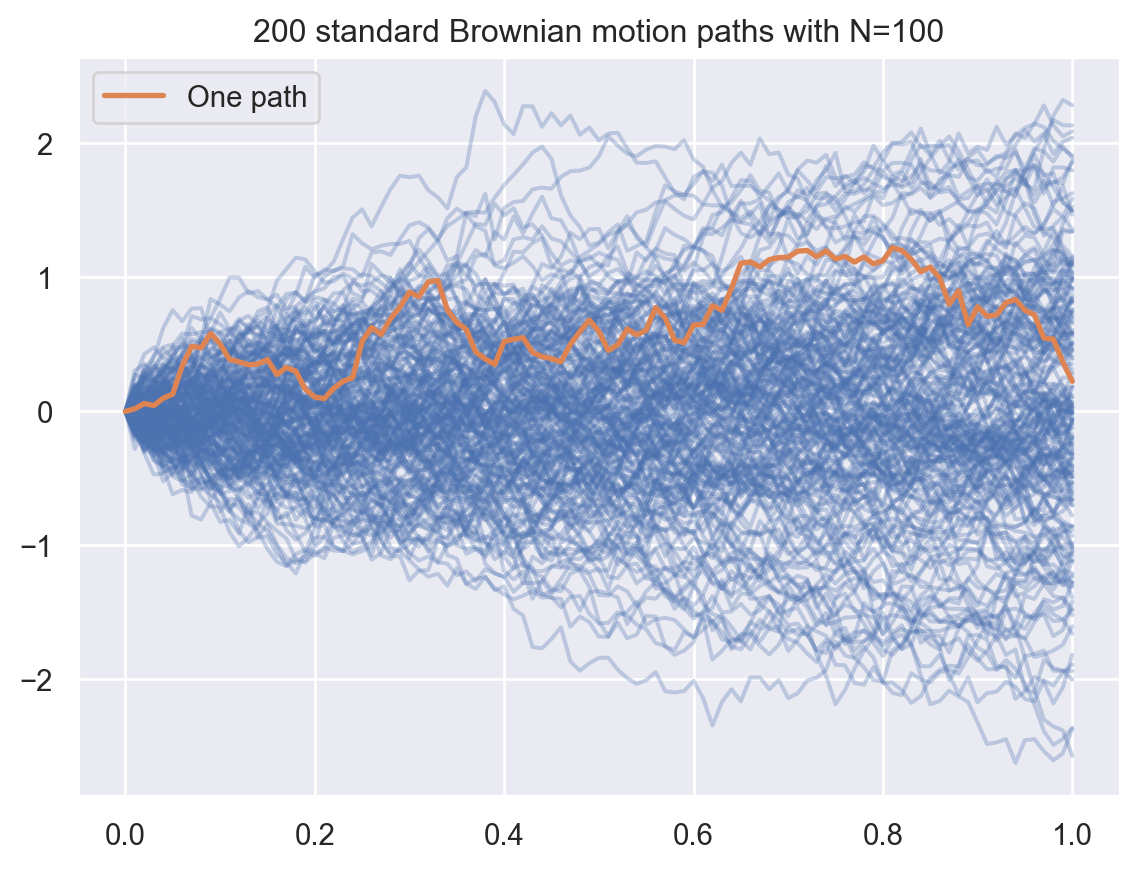
D’après la définition 2. on a \[ \begin{pmatrix} B_{t_1} \\ \vdots \\ B_{t_N} \end{pmatrix} \sim \mathcal{N} \big(0, \Sigma \big), \quad \text{avec} \quad \Sigma = \begin{pmatrix} t_1 & t_1 & \cdots & t_1 & t_1 \\ t_1 & t_2 & \cdots & t_2 & t_2 \\ \vdots & \vdots & \ddots & \ddots & \vdots \\ t_1 & t_2 & \cdots & t_{N-1} & t_{N-1} \\ t_1 & t_2 & \cdots & t_{N-1} & t_N \end{pmatrix}. \]
La matrice \(\Sigma\) est symétrique définie positive donc par la décomposition de Cholesky il existe une matrice triangulaire inférieure \(L\) telle que \(L L^\top = \Sigma\). Ainsi le vecteur \((B_{t_1}, \dots, B_{t_N})\) a même loi que \(L G\) où \(G = (G_1, \dots, G_n) \sim \mathcal{N}(0, \operatorname{Id}_N)\).
Pour tout vecteur aléatoire \(X \in \mathbf{L}^2(\mathbf{R}^d)\), et toute matrice \(A \in \mathcal{M}(q, d)\), le vecteur \(Y = AX \in \mathbf{L}^2(\mathbf{R}^q)\) et la matrice de covariance du vecteur \(Y\) est donnée par \(C_Y = A C_X A^\top\) où \(C_X\) est la matrice de covariance du vecteur \(X\).
gaussian_process_1ddef gaussian_process_1d(n_paths: int,
times: np.ndarray,
means: np.ndarray,
covariances: np.ndarray,
rng: np.random.Generator = None) -> np.ndarray:
"""
Simule des trajectoires de mouvement brownien standard en 1D.
Args:
n_paths: Nombre de trajectoires à simuler.
times: Tableau 1d (list, tuple ou np.ndarray) de taille `n_times`
qui contient les instants $t_k$.
means: Tableau 1d (list, tuple ou np.ndarray) de taille `n_times`
qui contient les moyennes aux instants $t_k$.
covariances: `np.ndarray` de forme `(n_times-1, n_times-1)`
qui contient les covariances
rng: objet `Generator`, création si `None` (non recommandé).
Returns:
np.ndarray de forme `(n_paths, n_times)` contenant les trajectoires
aux instants $t_k$ du processus gaussien.
"""
# Initialisation du générateur pseudo-aléatoire si non fourni
if rng is None:
rng = np.random.default_rng()
L = np.linalg.cholesky(covariances)
G = rng.standard_normal((n_paths, len(times)-1))
B = np.einsum('ij,pj->pi', L, G)
# ou bien B = (L @ G.T).T
paths = means + np.hstack([np.zeros((n_paths,1)), B])
return pathsN, M = 100, 200
T = 1
times = np.arange(N+1)*(T / N)
Sigma = np.minimum(times[1:], times[1:,np.newaxis])
paths = gaussian_process_1d(M, times,
means = np.zeros_like(times),
covariances = Sigma,
rng = rng)
fig, ax = plt.subplots()
for path in paths:
ax.plot(times, path, color='C0', alpha=0.3)
ax.plot(times, paths[0,:], color='C1', lw=2, label='One path')
ax.legend()
ax.set_title(f"{M} standard Brownian motion paths with N={N}");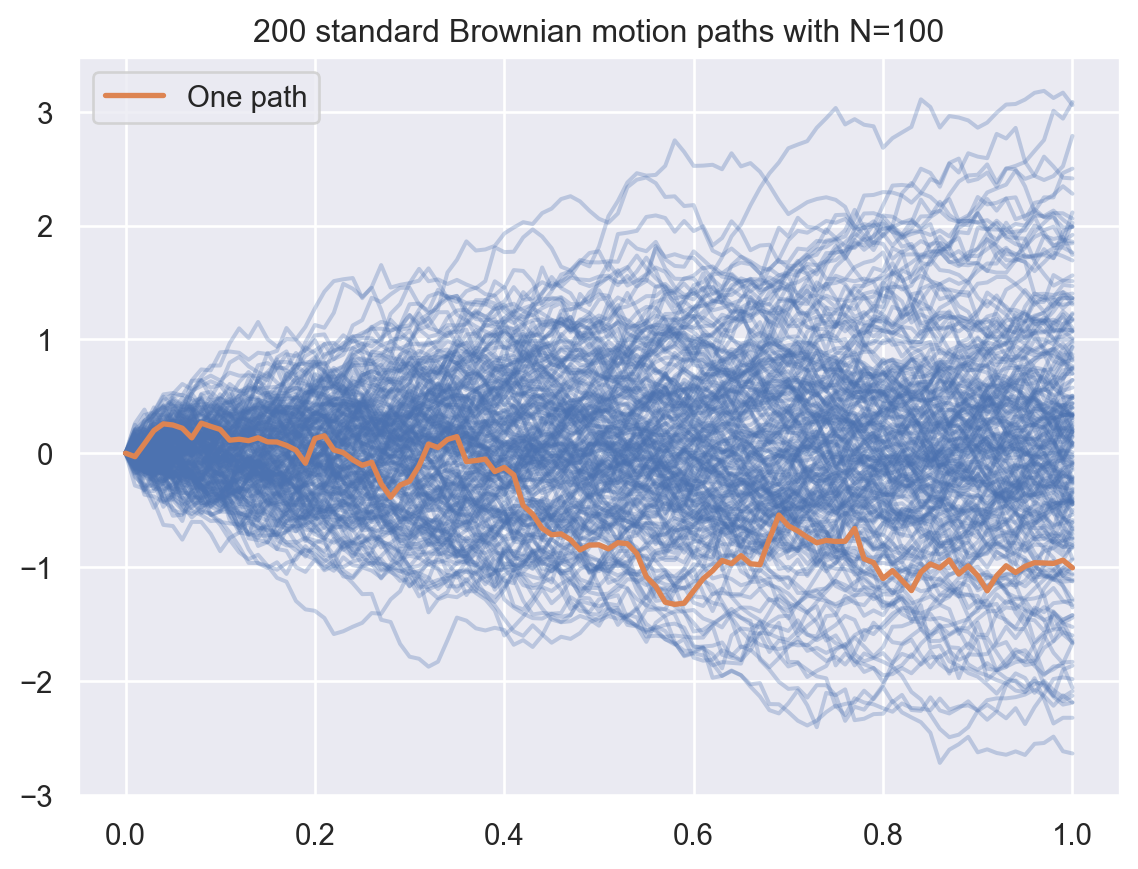
La construction trajectorielle de Paul Lévy permet aussi de simuler le mouvement Brownien: il s’agit d’affiner la trajectoire, itération après itération, en suivant le découpage dyadique de \([0,1]\). Pour cela il faut déterminer la loi conditionnelle du Brownien à l’instant \(t\) sachant ses positions \(x\) et \(y\) respectivement en des instants \(l\) et \(r\) tels que \(l < t < r\).
La propriété principale est la suivante \(\forall l < t < r\), \(\forall x, y \in\mathbf{R}\), \[ B_t \big\lvert \{B_l = x, B_r = y \} \sim \mathcal{N}\left(\frac{r-t}{r-l} x + \frac{t-l}{r-l} y, \frac{(r-t)(t-l)}{r-l} \right). \] En particulier si on considère un temps \(t = \frac{l+r}{2}\) (milieu de \([l,r]\)) alors \(\forall x, y \in\mathbf{R}\), \[ B_{\frac{l+r}{2}} \big\lvert \{B_l = x, B_r = y \} \sim \mathcal{N}\left(\frac{x+y}{2}, \frac{r-l}{4} \right). \]
Soit \((G_{k,n}, 0 \le k < 2^n, n \ge 0)\) une suite (doublement indicée) i.i.d. de loi \(\mathcal{N}(0,1)\). On introduit les instants dyadiques de \([0,1]\) par la notation \(t_k^n = \frac{k}{2^n}\) (on a alors \(t^{n+1}_{2k+1} = \frac{1}{2} (t^n_k + t^n_{k+1})\) les nouveaux instants qui apparaissent à l’échelle \(n+1\)). On définit une suite de processus \((X^n_t)_{t \in [0,1]}\) où pour \(n\) fixé \((t \mapsto X^n_t)\) est linéaire sur chaque intervalle \([t^n_k, t^n_{k+1}[\), et aux points \(t_k^n\): \[ \begin{cases} X^0_0 = 0, \quad \text{et} \quad X^0_1 = G_{0,0}, \\ X^{n+1}_{t^n_k} = X^n_{t^n_k}, \quad \text{et} \quad X^{n+1}_{t^{n+1}_{2k+1}} = X^{n}_{t^{n+1}_{2k+1}} + 2^{-\frac{n}{2}-1} G_{2k+1, n+1}. \end{cases} \] Noter que \(X^n_{t^{n+1}_{2k+1}} = \frac{1}{2} \big(X^n_{t^n_k} + X^n_{t^n_{k+1}}\big)\) par la construction linéaire par morceaux.
refine_brownian_1ddef refine_brownian_1d(times: np.ndarray, paths: np.ndarray,
rng: np.random.Generator=None) -> tuple :
""" Raffine les trajectoires Browniennes et les instants de temps
en doublant la résolution temporelle.
Args:
times: np.ndarray de taille N qui contient les instants $t_k$.
paths: np.ndadday de forme (N, M) où
- N est le nombre de points temporels
- M est le nombre de trajectoires simulées.
rng: objet `Generator`, création si `None` (non recommandé).
Returns:
tuple:
- np.ndarray de forme (2*N - 1, ) avec les instants interpolés.
- np.ndarray de forme (M, 2*N - 1) avec les trajectoires raffinées.
"""
if rng is None:
rng = np.random.default_rng()
M, N = paths.shape
dt = np.diff(times)
# Raffinement des instants de temps
r_times = np.empty(2*N - 1)
r_times[0::2] = times # Copie des instants originaux
r_times[1::2] = (times[:-1] + times[1:]) / 2 # Interpolation
# Raffinement des trajectoires
r_paths = np.empty((M, 2*N - 1))
r_paths[:, 0::2] = paths
r_paths[:, 1::2] = (paths[:, :-1] + paths[:, 1:]) / 2 \
+ np.sqrt(dt / 4) * rng.standard_normal((M, N-1))
return r_times, r_pathst0 = np.array([0, 1])
W0 = brownian_1d(M, t0, rng=rng)
## Exemple d'appel de la fonction
t1, W1 = refine_brownian_1d(t0, W0, rng=rng)
## Création d'un dictionnaire pour les résultats
result = {
0: (t0, W0),
1: (t1, W1),
}
## On complète le dictionnaire des résultats
for n in range(2, 5):
result[n] = refine_brownian_1d(*result[n-1], rng=rng)
## On fait un tracé des trajectoires en fonction du niveau de raffinement $n$
fig, axs = plt.subplots(ncols=2, nrows=2, figsize=(8,8),
sharex=True, sharey=True, layout='tight')
for n, ax in enumerate(axs.flat):
t, paths = result[n+1]
for path in paths:
ax.plot(t, path, color='C0', alpha=0.3)
ax.plot(t, paths[0], color='C1', lw=2, label=f'One path')
ax.legend()
ax.set_title(f'Iteration $n$={n+1}')
fig.suptitle(f'Raffinement des trajectoires en fonction du paramètre $n$');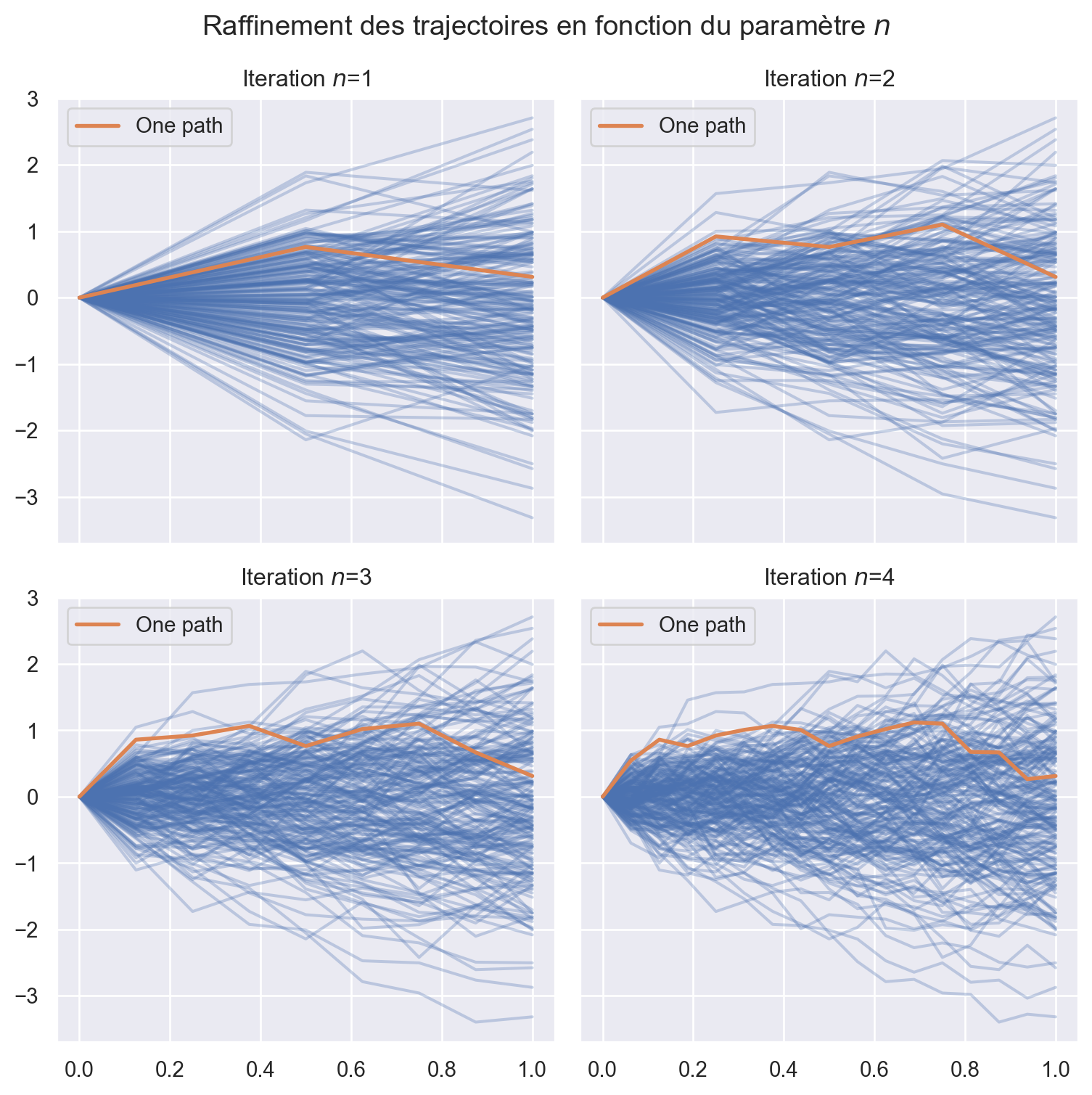
On considère maintenant un mouvement Brownien en dimension 2, \(B = (B^1, B^2)\) de corrélation \(\rho \in [-1,1]\), c’est à dire tel que \(\operatorname{Corr}(B^1_t, B^2_t) = \rho t\). On rappelle que \(B\) se représente sous la forme \[ \begin{cases} B^1_t &= W^1_t, \\ B^2_t &= \rho W^1_t + \sqrt{1-\rho^2} W^2_t \end{cases} \] où \(W = (W^1, W^2)\) est un mouvement Brownien bidimensionnel dont les composantes sont indépendantes.
brownian_2ddef brownian_2d(n_paths: int,
times: np.ndarray,
correlation: float = 0.,
increments: bool = False,
rng: np.random.Generator = None) -> np.ndarray:
"""
Simule des trajectoires de mouvement brownien standard en 2D, avec
option de corrélation entre les 2 dimensions.
Args:
n_paths: Nombre de trajectoires à simuler.
times: tableau 1d (list, tuple ou np.ndarray) de taille `n_times`
qui contient les instants $t_k$.
correlation: Valeur du paramètre de corrélation $\rho \in [-1,1]$.
increments: Si `True`, les accroissements sont retournés,
sinon ce sont les trajectoires.
rng: objet `Generator`, création si `None` (non recommandé).
Returns:
- si `increments=False`: np.ndarray de forme `(n_paths, n_times, 2)`
contenant les trajectoires aux instants $t_k$.
- si `increments=True`: np.ndarray de forme `(n_paths, n_times-1, 2)`
contenant les accroissements entre les instants $t_k$.
"""
# Initialisation du générateur pseudo-aléatoire si non fourni
if rng is None:
rng = np.random.default_rng()
# Calcul des accroissements avec corrélation
dt = np.diff(times)
dW = np.sqrt(dt)[None,:,None] * rng.standard_normal((n_paths, len(dt), 2))
dB = np.empty_like(dW)
dB[:,:,0] = dW[:,:,0]
dB[:,:,1] = correlation * dW[:,:,0] \
+ np.sqrt(1-correlation**2) * dW[:,:,1]
# Si les accroissements sont demandés
if increments:
return dB
# Construction des trajectoires browniennes (cumul des incréments)
paths = np.hstack([np.zeros((n_paths,1,2)), np.cumsum(dB, axis=1)])
return paths<>:14: SyntaxWarning: "\i" is an invalid escape sequence. Such sequences will not work in the future. Did you mean "\\i"? A raw string is also an option.
<>:14: SyntaxWarning: "\i" is an invalid escape sequence. Such sequences will not work in the future. Did you mean "\\i"? A raw string is also an option.
/var/folders/pr/88jy049n5zn69sd1z3v73yvm0000gn/T/ipykernel_439/414599337.py:14: SyntaxWarning: "\i" is an invalid escape sequence. Such sequences will not work in the future. Did you mean "\\i"? A raw string is also an option.
correlation: Valeur du paramètre de corrélation $\rho \in [-1,1]$.N, M = 100, 200
T = 1
times = np.linspace(0, T, N)
## imes = np.arange(N+1)*(T / N)
rho = -0.7
paths = brownian_2d(M, times, correlation=rho)
## ns.lineplot(x=times, y=W2[0,:,0], color='C0', label='Dimension 1')
## ns.lineplot(x=times, y=W2[0,:,1], color='C1', label='Dimension 2')
fig, ax = plt.subplots()
for path in paths:
ax.plot(path[:,0], path[:,1], color='C0', alpha=0.2)
ax.plot(paths[0,:,0], paths[0,:,1], color='C1', lw=2, label='One path')
ax.legend()
ax.set_title(fr"{M} 2d-Brownian motion paths with N={N}, $\rho = {rho}$");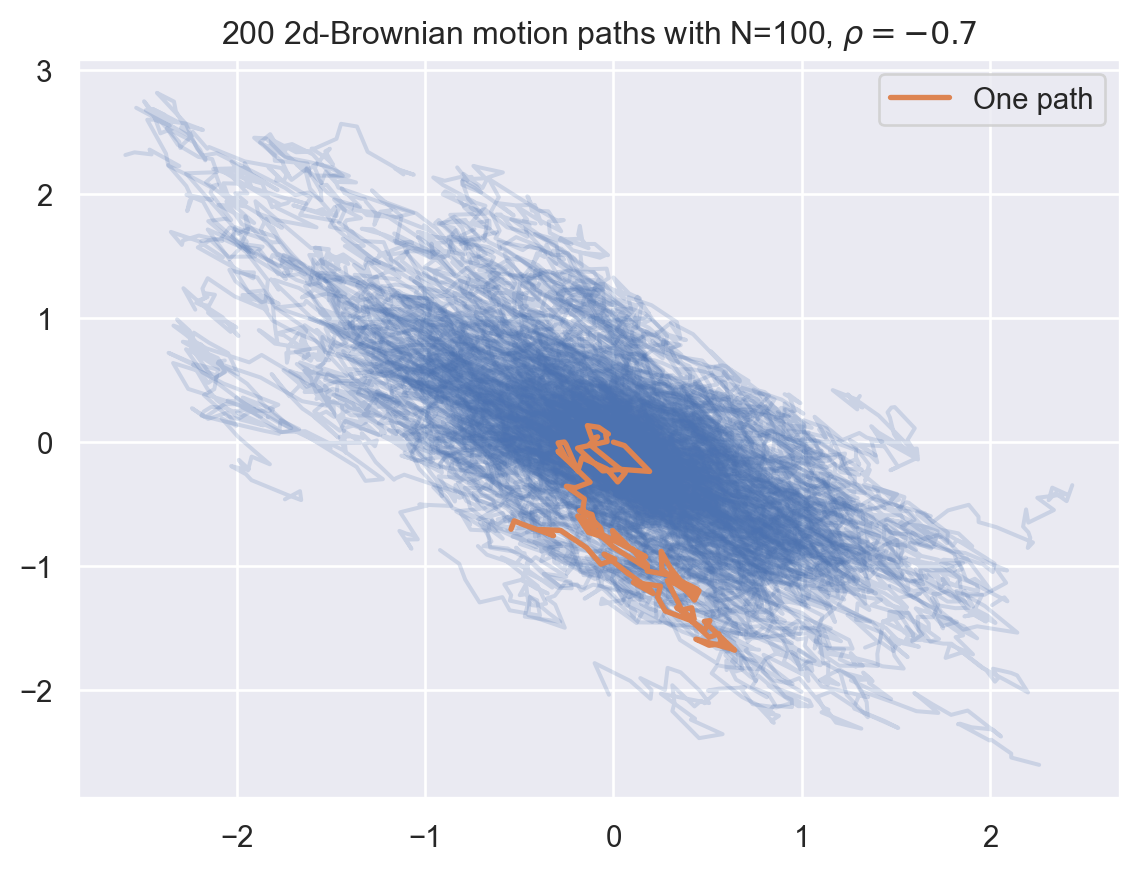
fig, ax = plt.subplots()
ax.plot(times, paths[0,:,0], color='C1', lw=2, ls='--', label='Dimension 1')
ax.plot(times, paths[0,:,1], color='C1', lw=2, ls=':', label='Dimension 2')
ax.legend()
ax.set_title(fr"A path as a function of time, $\rho = {rho}$");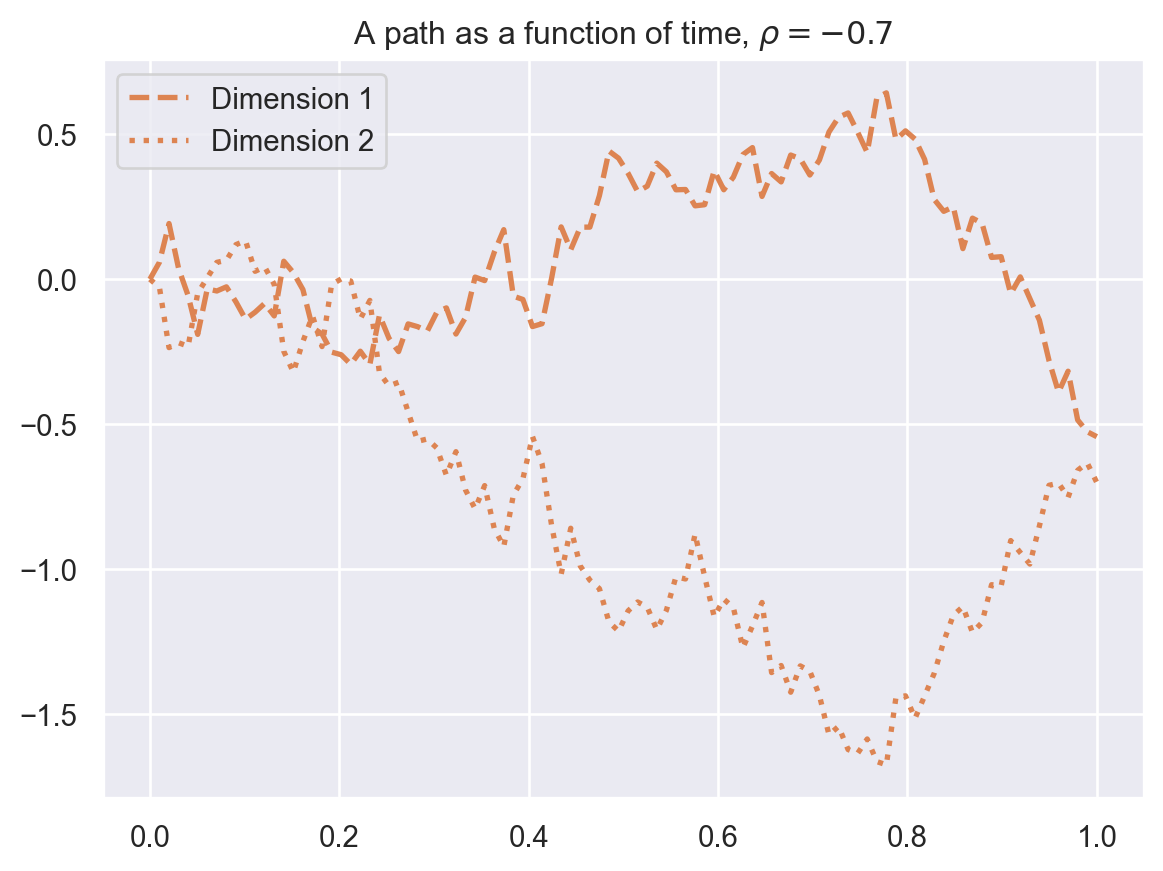
Exercice: écrire un code pour vérifier que la corrélation empirique à chaque instant est bien “égale” à \(\rho = -0.7\). Vous pouvez faire 2 codes:
mean et std et en agissant sur les bons axesnp.corrcoef (lire la documentation): pour agir sur le temps il faut faire une boucleW0, W1 = paths[:,1:,0], paths[:,1:,1]
## Solution à la main sans boucle
cov_t = np.mean((W0 - W0.mean(axis=0)) * (W1 - W1.mean(axis=0)), axis=0)
corr_t = cov_t / (W0.std(axis=0) * W1.std(axis=0))
## Solution en utilisant np.corrcoef
corr_t = [np.corrcoef(W0[:, t], W1[:, t])[0, 1] for t in range(W0.shape[1])]
fig, ax = plt.subplots()
ax.plot(times[1:], corr_t, color='C2', lw=2, label='Empirical correlation')
ax.hlines(rho, times[1], times[-1], color='C3', label='True correlation')
ax.legend()
ax.set_title(fr"Empirical correlation as a function of time, $\rho = {rho}$");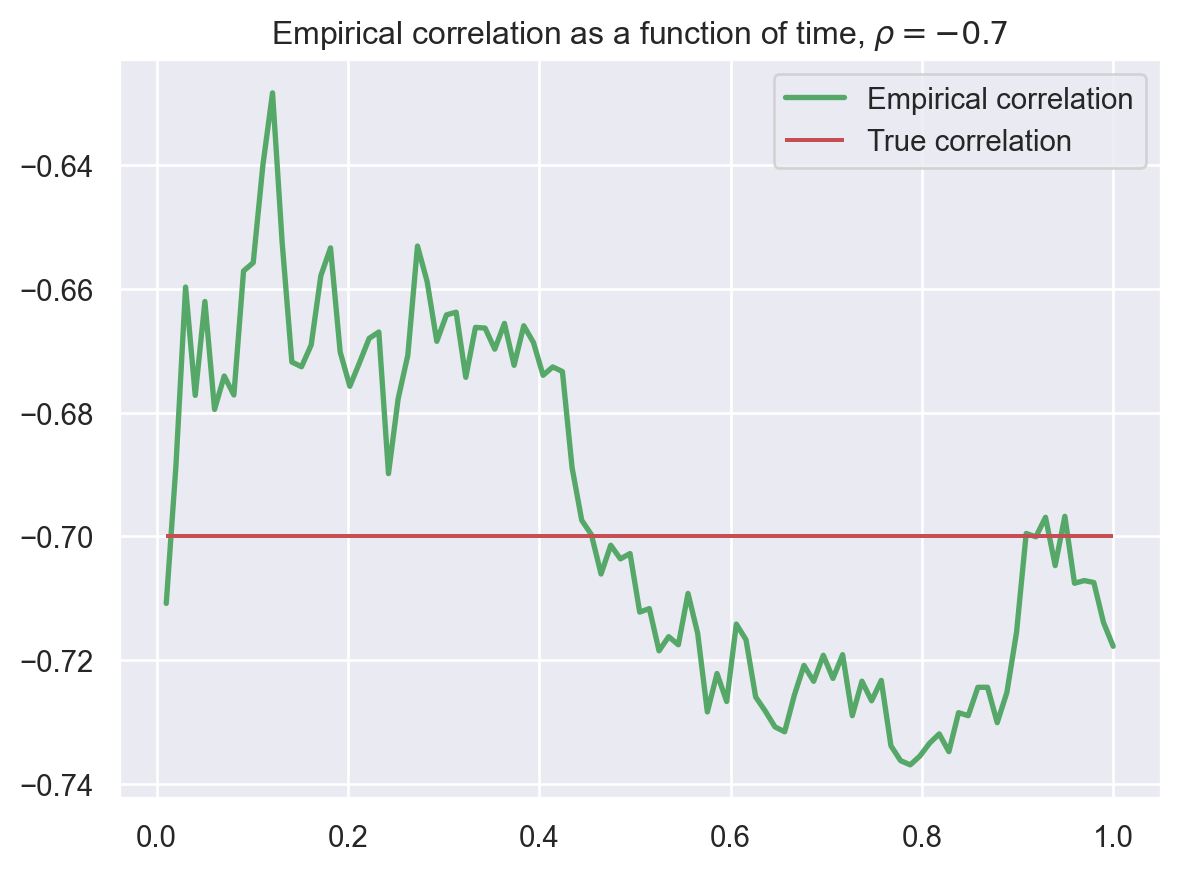
Dans le tracé précédent il faudrait rajouter l’intervalle de confiance autour de chaque point de corrélation empirique!
On suppose que la matrice de corrélation entre les dimensions est donnée par \(\Sigma\) une matrice symétrique définie positive.
Exercice: écrire le code de la fonction brownian_md, vous pouvez faire la simulation à la main en utilisant la transormation de Cholesky de Sigma ou bien utiliser multivariate_normal (cf. doc).
Modèle classique pour représenter le cours d’un actif financier.
On considère le processus \(S = (S_t)_{t \in [0,T]}\) solution de l’EDS sur \([0,T]\) \[ \operatorname{d}\!S_t = r S_t \operatorname{d}\!t + \sigma S_t \operatorname{d}\!B_t, \quad S_0 = x, \] c’est à dire (cf. cours calcul stochastique), pour tout \(t \in [0,T]\) \[ S_t = x \exp \bigl((r- \sigma^2/2) t + \sigma B_t \bigr). \]
black_scholes_1ddef black_scholes_1d(n_paths: int,
times: np.ndarray,
init_value: float,
r: float,
sigma: float,
rng: np.random.Generator = None) -> np.ndarray:
"""
Simule des trajectoires de mouvement brownien standard en 1D.
Args:
n_paths: Nombre de trajectoires à simuler.
times: tableau 1d (list, tuple ou np.ndarray) de taille `n_times`
qui contient les instants $t_k$.
init_value: valeur initiale de l'actif
r: taux d'intérêt
sigma: volatilité de l'actif
rng: objet `Generator`, création si `None` (non recommandé).
Returns:
- np.ndarray de forme `(n_paths, n_times)` contenant les
trajectoires aux instants $t_k$.
"""
Bt = brownian_1d(n_paths, times, rng=rng)
St = S0 * np.exp((r - 0.5*sigma**2) * times + sigma * Bt)
return StVoici un code illustrant l’utilisation de cette fonction.
T, N = 1, 100
times = np.arange(N+1)*(T / N)
S0, r, sigma = 100, 0.04, 0.20
M = 200
paths = black_scholes_1d(M, times, init_value=S0, r=r, sigma=sigma, rng=rng)
fig, ax = plt.subplots()
for path in paths:
ax.plot(times, path, color='C0', alpha=0.3)
ax.plot(times, paths[0,:], color='C1', lw=2, label='One path')
ax.legend()
ax.set_title(f"{M} standard Black-Scholes paths with N={N} " \
+ fr"($r={r}$, $\sigma={sigma}$)");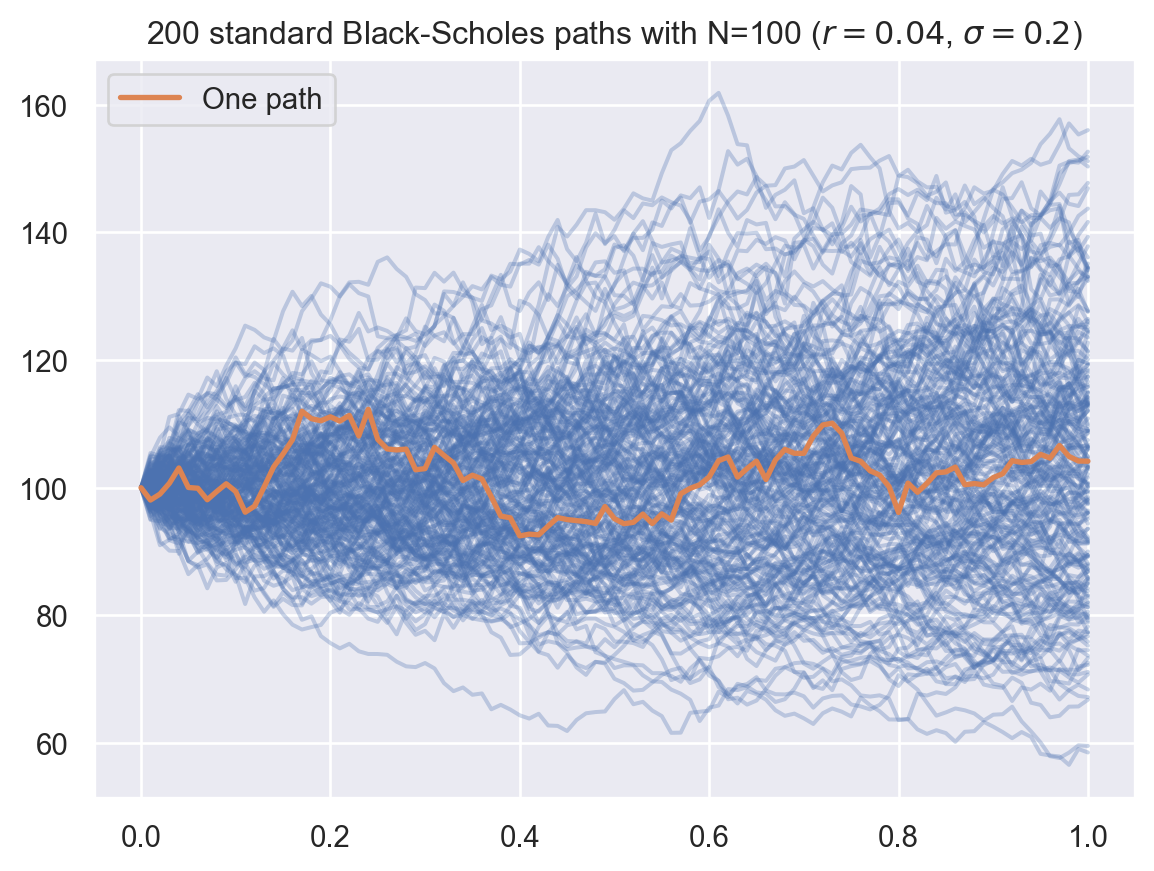
(cf. projet Options Basket à la fin de la séance).
Méthode numérique basée sur la loi des grands nombres et le théorème central limite pour donner un intervalle de confiance (asymptotique).
La seule valeur moyenne \(\bar I_M = \frac{1}{M} \sum_{k=1}^M X_k\) d’un échantillon \((X_1, \dots, X_M)\) n’est pas suffisante pour déterminer \(I = \mathbf{E}[X_1]\). Il faut toujours renvoyer la valeur de l’estimateur \(I_M\) et son intervalle de confiance \[
I \in \Bigl] \bar I_M- q_{1-\frac{\alpha}{2}} \frac{\sigma}{\sqrt{M}};
\bar I_M + q_{1 - \frac{\alpha}{2}} \frac{\sigma}{\sqrt{M}}
\Bigr[ \quad \text{avec probabilité $p = 1-\alpha \in ]0,1[$}
\] où \(q_{1-\frac{\alpha}{2}}\) est le quantile de niveau \(1-\frac{\alpha}{2}\) de la loi normale centrée réduite et \(\sigma^2 = \operatorname{var}(X_1)\).
Pour \(\alpha = 0.05\) c’est à dire \(p = 0.95\) on a \(1-\frac{\alpha}{2} = 1.96\).
La moyenne seule ne suffit pas, il faut toujours donner l’intervalle de confiance.
monte_carlodef monte_carlo(sample: np.ndarray, proba: float = 0.95) -> dict:
"""
Calcule l'estimation Monte Carlo de la moyenne d'un échantillon, et
fournit un intervalle de confiance pour la moyenne.
Args:
- sample (np.ndarray): Un tableau contenant les échantillons à analyser.
- proba (float): Niveau de confiance pour l'intervalle (défaut: 0.95).
Returns:
- dict: Un dictionnaire contenant les valeurs suivantes :
- "mean" (float): Moyenne de l'échantillon.
- "var" (float): Variance de l'échantillon.
- "lower" (float): Borne inférieure de l'intervalle de confiance.
- "upper" (float): Borne supérieure de l'intervalle de confiance.
"""
# Moyenne et variance de l'échantillon
mean = np.mean(sample)
var = np.var(sample, ddof=1)
# Calcul de l'intervalle de confiance pour la moyenne
alpha = 1 - proba
quantile = sps.norm.ppf(1 - alpha / 2)
ci_size = quantile * np.sqrt(var / sample.size)
return {
"mean": mean,
"var": var,
"lower": mean - ci_size,
"upper": mean + ci_size
}Exercice: Calculer le prix d’un Call dans un modèle de Black-Scholes par sa formule fermée et comparer le prix Monte Carlo pour différentes valeurs de \(M\).
S0, r, sigma = 100, 0.04, 0.2
K = 100
times = np.array([0, 1])
results = {}
Ms = 10**np.arange(3, 8)
for M in Ms:
St = black_scholes_1d(M, times, init_value=S0, r=r, sigma=sigma)
payoffs = np.exp(-r*times[-1]) * np.maximum(St[:,-1] - K, 0)
results[M] = monte_carlo(payoffs)
import pandas as pd
pd.DataFrame(results)| 1000 | 10000 | 100000 | 1000000 | 10000000 | |
|---|---|---|---|---|---|
| mean | 9.966109 | 9.676087 | 9.916655 | 9.909553 | 9.925093 |
| var | 206.303580 | 194.807909 | 209.727308 | 207.766515 | 207.840395 |
| lower | 9.075881 | 9.402527 | 9.826897 | 9.881302 | 9.916157 |
| upper | 10.856338 | 9.949646 | 10.006414 | 9.937804 | 9.934028 |
black_scholes_calldef black_scholes_call(S0: float, K: float, T: float,
r: float, sigma: float) -> float:
"""
Calcule le prix d'un Call européen dans le modèle de Black-Scholes.
Args:
- S0 (float): Prix actuel de l'actif sous-jacent.
- K (float): Prix d'exercice du Call.
- T (float): Temps jusqu'à l'échéance en années.
- r (float): Taux d'intérêt sans risque.
- sigma (float): Volatilité du sous-jacent.
Returns:
- float: Prix du Call européen.
"""
# Calcul des paramètres d1 et d2
d1 = (np.log(S0 / K) + (r + 0.5 * sigma ** 2) * T) / (sigma * np.sqrt(T))
d2 = d1 - sigma * np.sqrt(T)
# Calcul du prix du Call
call_price = S0 * sps.norm.cdf(d1) - K * np.exp(-r * T) * sps.norm.cdf(d2)
return call_priceprice = black_scholes_call(S0, K, T=1, r=r, sigma=sigma)
print("Prix par formule fermée:", price)Prix par formule fermée: 9.925053717274437On considère \(d \ge 2\) actifs financiers dont la loi à l’instant \(T > 0\) est modélisée par une loi log-normale c’est à dire \[ \forall i \in \{1,\dots,d\}, \quad S^i_T = S^i_0 \exp\Bigl( \bigl(r-\frac{\sigma_i^2}{2}\bigr) T + \sigma_i \sqrt{T} \tilde G_i \Bigr) \] où le vecteur \((\tilde G_1,\dots, \tilde G_d)\) est gaussien centré de matrice de corrélation \(\Sigma\) et les constantes \(r > 0\), \(\sigma_i > 0\) sont fixées. Il s’agit d’actifs financiers \((S^i_t)_{t \in [0,T]}\), \(1 \le i \le d\), modélisés par un processus de Black-Scholes multidimensionnel. On introduit la matrice \(L\) triangulaire inférieure obtenue par la décomposition de Cholesky de la matrice \(\Sigma = L L^\top\).
A l’aide de cette matrice \(L\), on définit la fonction \(\Phi:\mathbf{R}^d \to \mathbf{R}^d\) telle que \[ (S^1_T, \dots, S^d_T) = \Phi(G_1, \dots, G_d) \quad \text{ou encore} \quad S^i_T = \Phi_i(G_1, \dots, G_d) \] où \((G_1, \dots, G_d) \sim \mathcal{N}(0, I_d)\) (l’égalité précédente est à considérer en loi).
On s’intéresse au prix d’une option européenne (aussi appelé produit dérivé européen) sur le panier de ces \(d\) actifs financiers, c’est à dire qu’on veut calculer \[ e^{-rT} \mathbf{E} \bigl[ X \bigr] \quad \text{avec} \quad X = \biggl(\frac{1}{d} \sum_{i=1}^d S^i_T - K\biggr)_+ \]
Implémenter un estimateur Monte Carlo pour calculer au centime près le prix pour différentes valeurs de \(K\), \(K \in \{80, 90, 100, 110, 120\}\).
Implémenter les méthodes de réduction de variance suivantes, et comparer les temps de calcul et la complexité: