5.3 TP – Modularité
Illustrons d’abord l’algorithme de Girvan-Newman et la sélection du nombre de cluster basée sur la modularité sur un exemple.
On travaille avec la library igraph et on utilisera les fonctions suivantes:
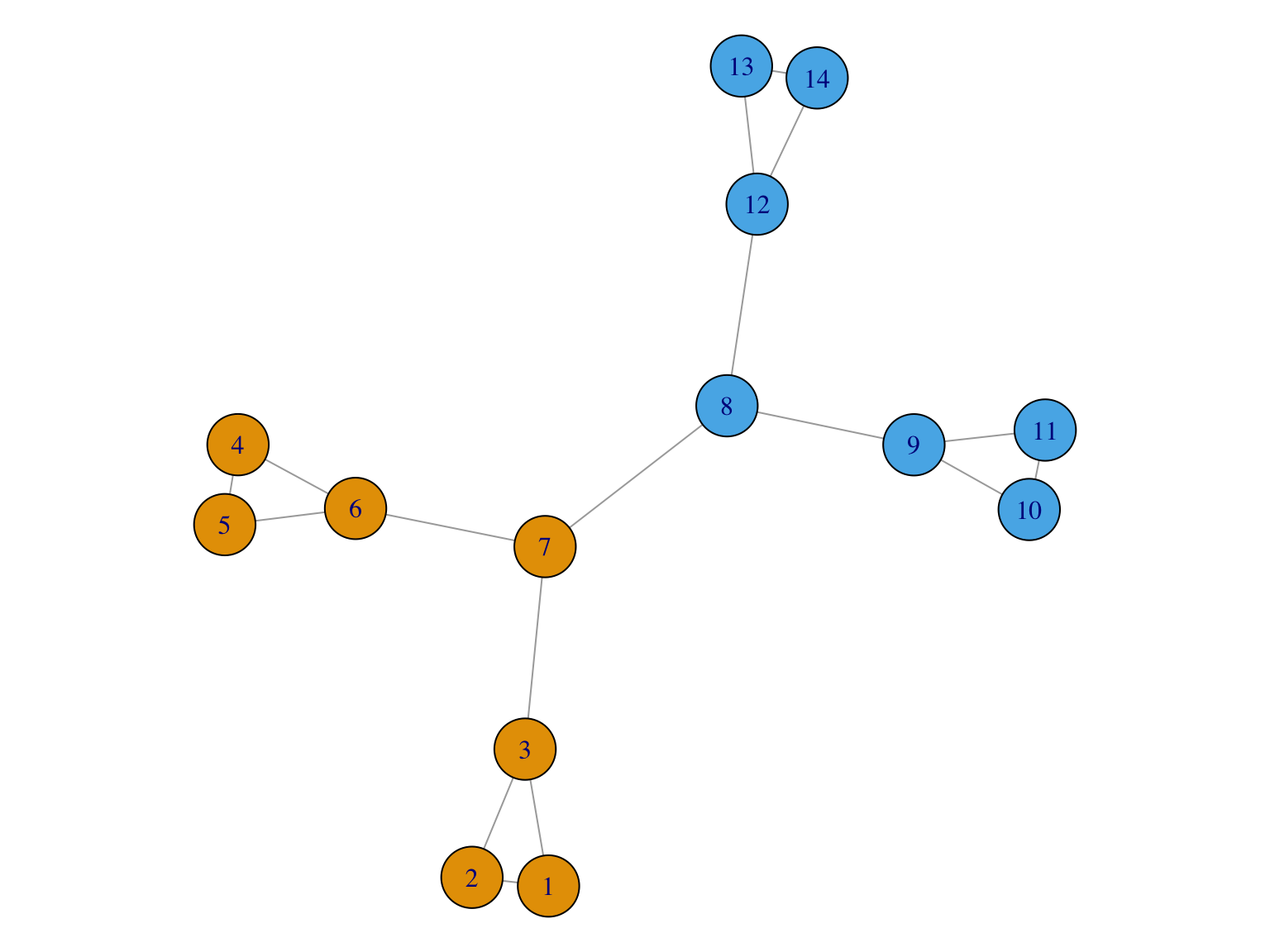
On construit le petit graphe suivant:
par(mar=c(0,0,0,0))
G1 <- make_full_graph(3)
G2 <- make_full_graph(3)
G3 <- make_full_graph(2)
G4 <- make_full_graph(3)
G5 <- make_full_graph(3)
G <- G1+G2+G3+G4+G5
G <- add.edges(G, c(6,7))
G <- add.edges(G, c(3,7))
G <- add.edges(G, c(8,9))
G <- add.edges(G, c(8,12))
plot(G)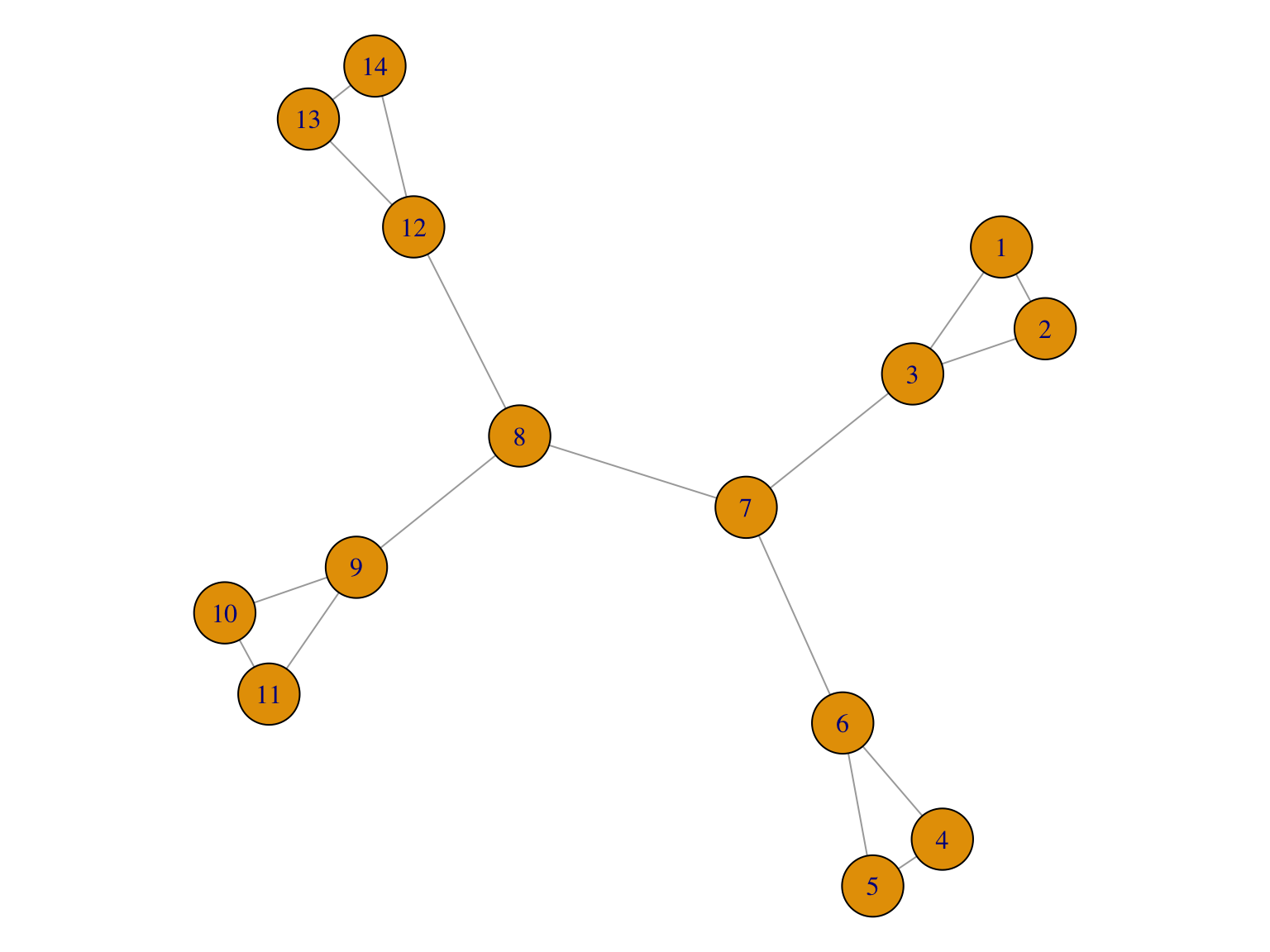
## [1] 1 12 12 1 12 12 49 12 12 1 12 12 1 33 33 33 33## + 17/17 edges from 58696dc:
## [1] 1-- 2 1-- 3 2-- 3 4-- 5 4-- 6 5-- 6 7-- 8 9--10 9--11 10--11
## [11] 12--13 12--14 13--14 6-- 7 3-- 7 8-- 9 8--12L’intermédiarité est la plus élevée pour la 7ème arête (avec une valeur de 49). C’est l’arête 7-8.
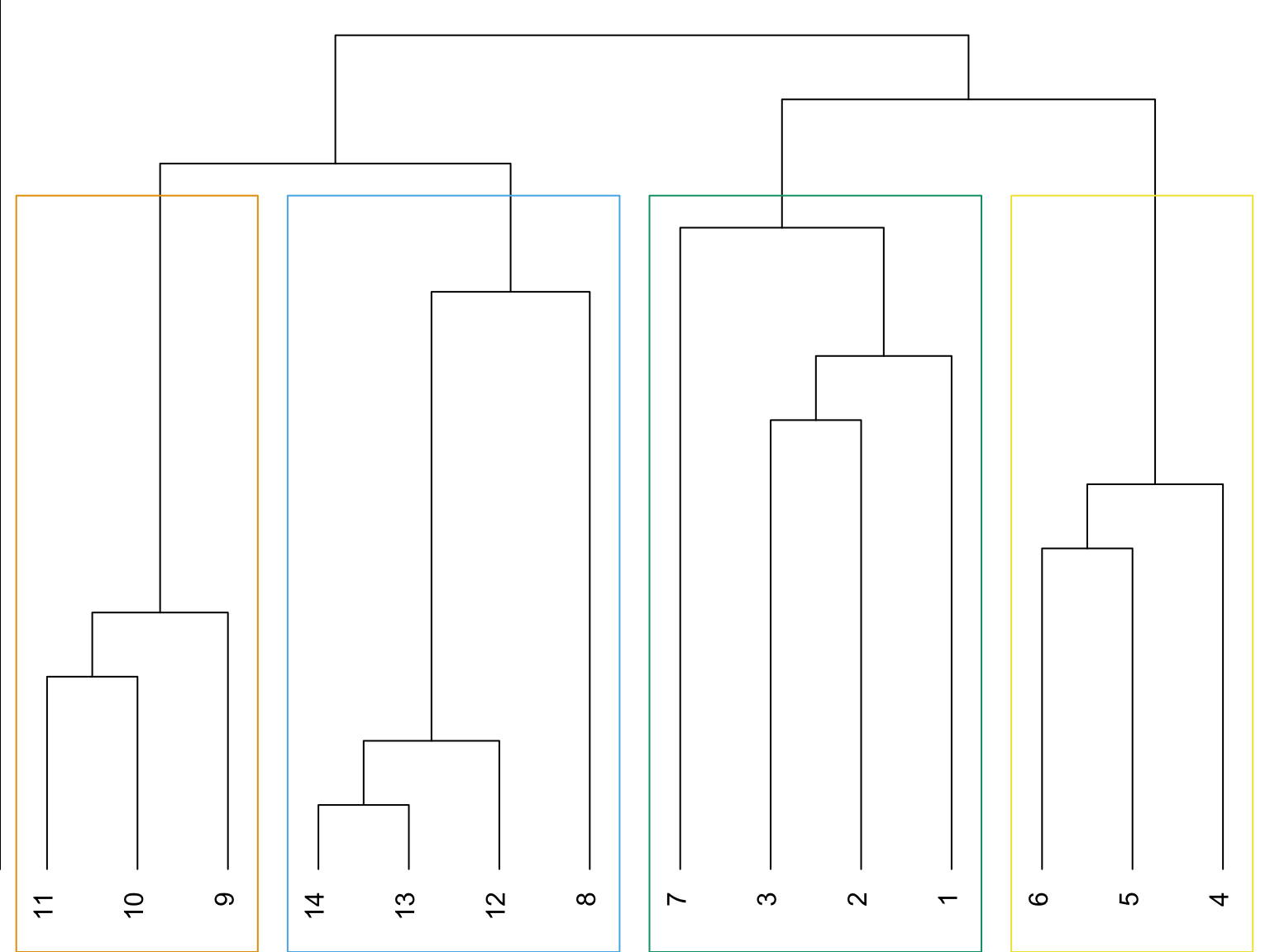
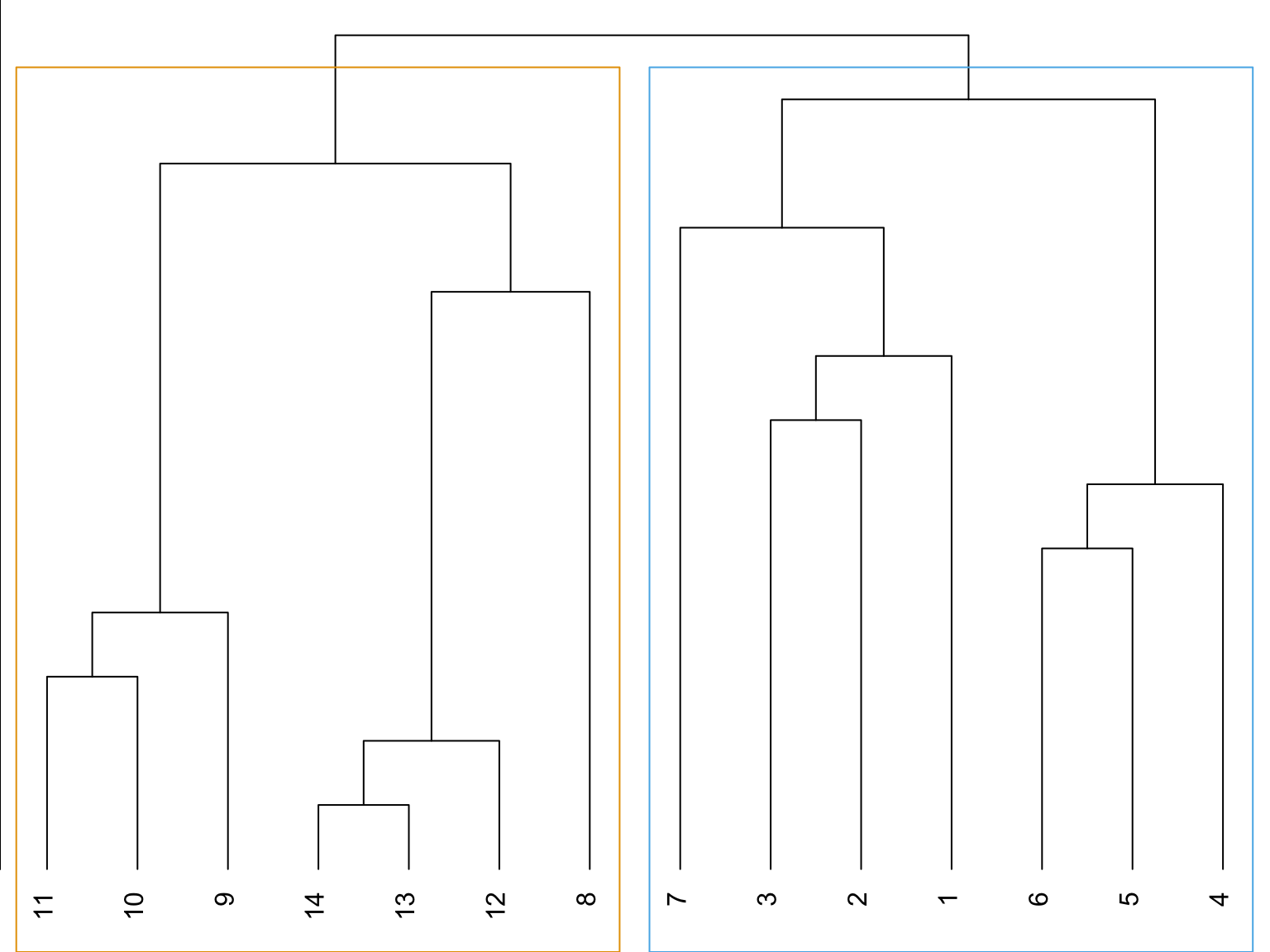
L’algorithme de Grivan-Newman a retiré l’arête 7-8: ce qui donne les clusters 1-7 et 8-14 Puis il a retiré l’arêtes 6-7: ce qui donne les clusters 4-5-6; 1-2-3-7 et 8-14…
Choix du nombre de clusters
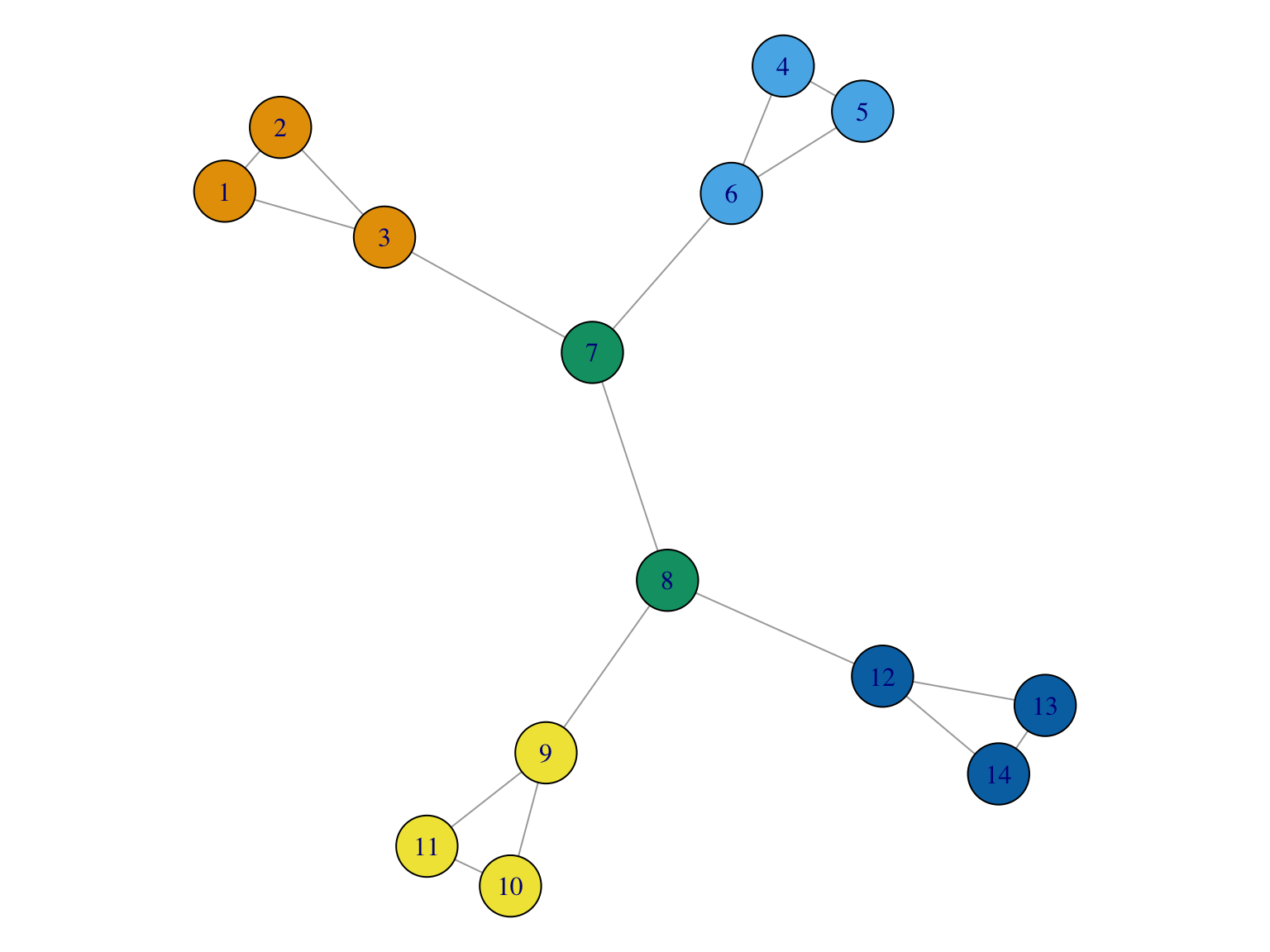
Le choix du nombre de clusters est basé sur la modularité. Ici on choisit 4 clusters:
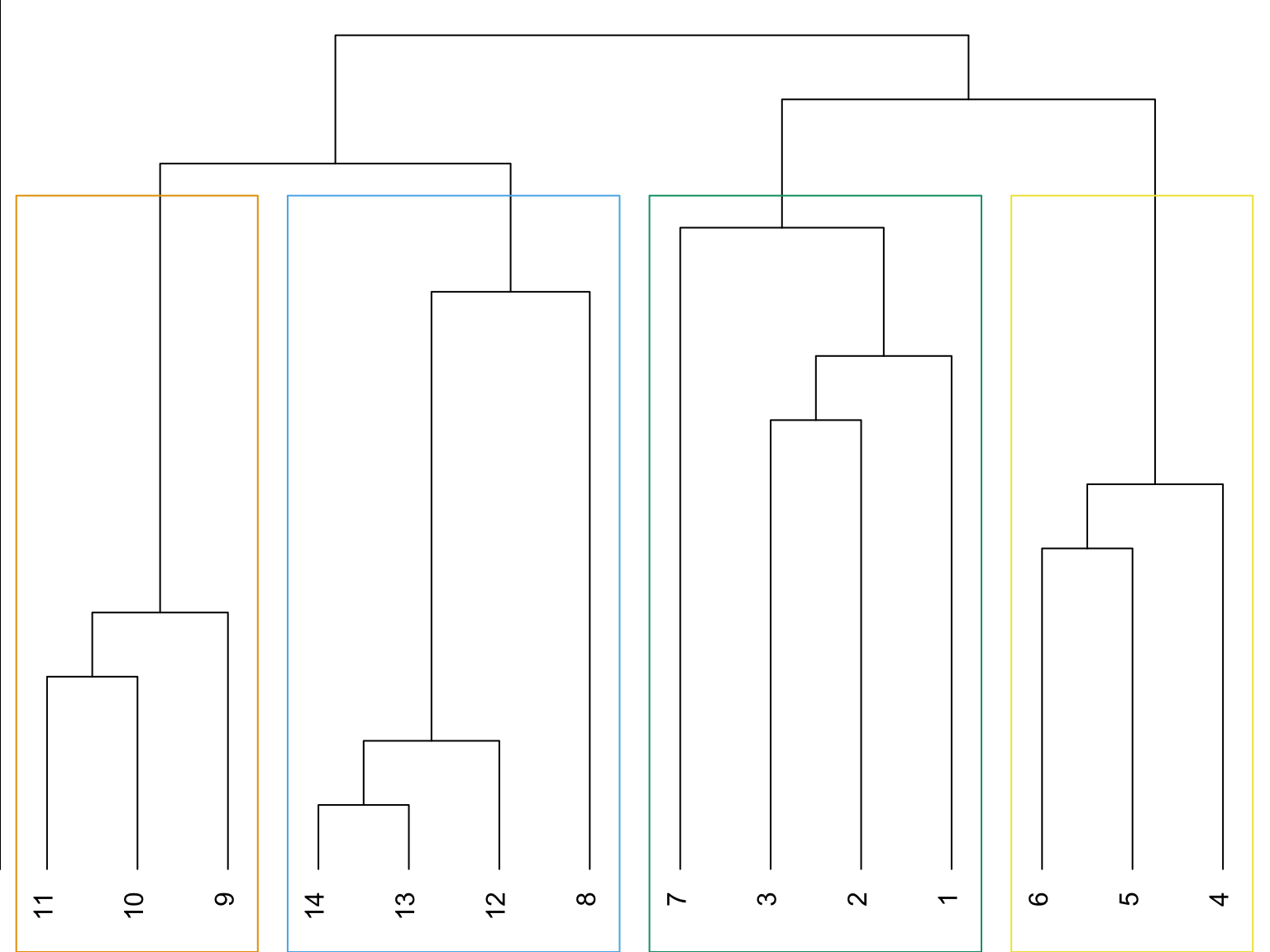
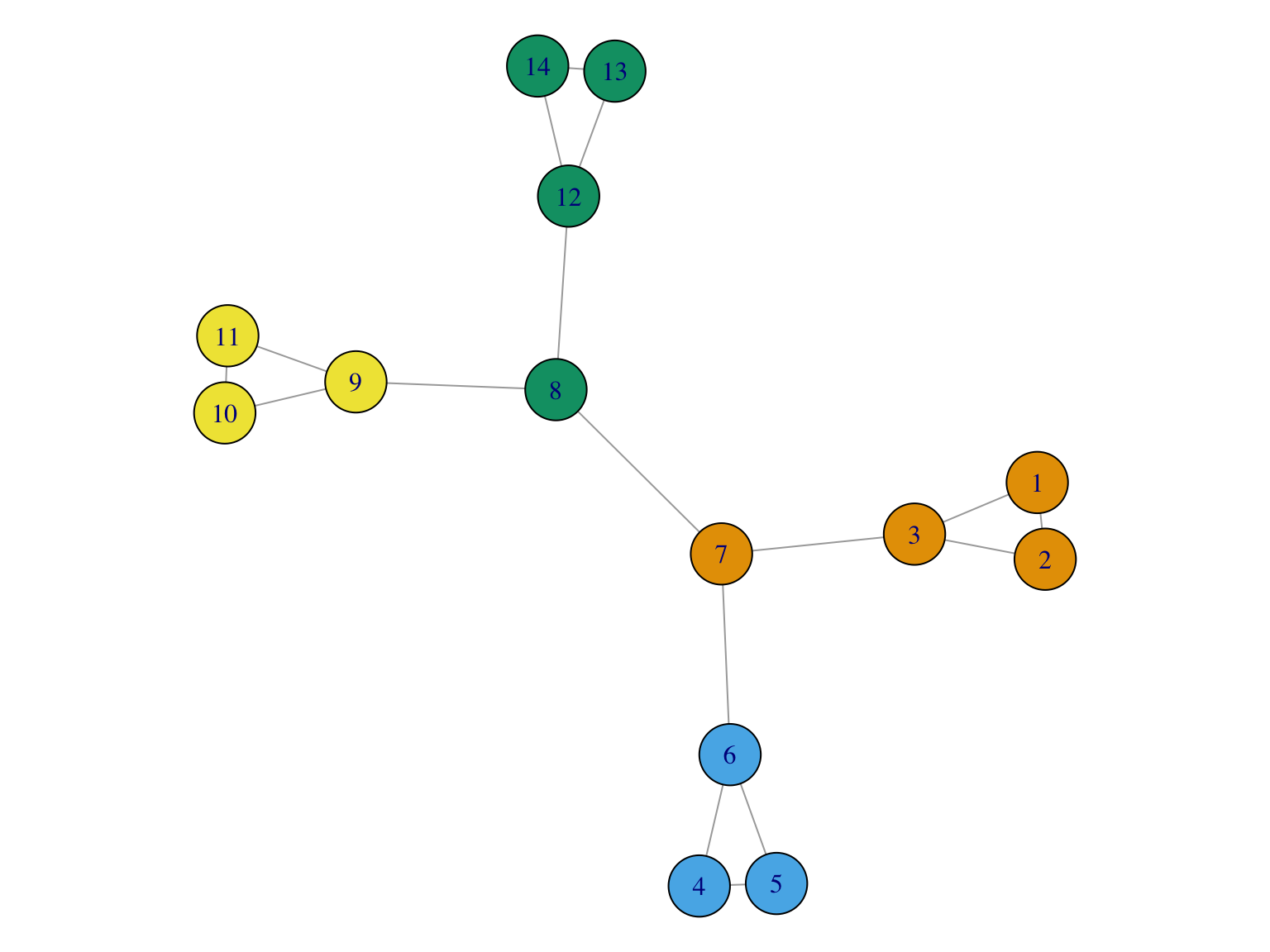
On peut choisir le nombre de clusters nous-même (coupure dans le dendogramme):
Algorithme de Louvain
Exercice
Maintenant, à vous sur le graphe karaté et sur le graphe friends.
Attention, la fonction cluster_edge_betweenness n’accepte pas les arêtes valuées, donc préciser weights=NULL si besoin. Importer le graphe friends en non dirigé.